Application Note SpectraMax M5マルチモードマイクロプレートリーダーでの
IMAP FPキナーゼアッセイ
- 均質なFPアッセイはHTSに適したフォーマットを提供
- ロバストなZ' factor値による正確で再現性の高いデータ
- SoftMax Proソフトウェアで設定済みのプロトコル
PDF版(英語)
プロテインキナーゼは、多くの細胞内プロセスの制御において中心的な役割を担っています。近年、キナーゼはがんやその他多くの疾患に対する最も重要な創薬標的のひとつに浮上しています。モレキュラーデバイスのIMAP® テクノロジーは、幅広いキナーゼの迅速な非放射性アッセイを可能にし、アッセイ開発とハイスループットスクリーニングの両方に適しています。IMAPテクノロジーは、ナノ粒子上に固定化された金属配位錯体を介したリン酸の結合に基づいています。IMAP結合体がキナーゼ反応によって生成したリン酸化ペプチドに結合すると、ペプチドの分子運動が変化し、ペプチドに結合した蛍光ラベルの蛍光偏光が増加します(図1)。このホモジニアスなアッセイは簡便で、ペプチド基質の配列に関係なく多くのキナーゼに適用できます。
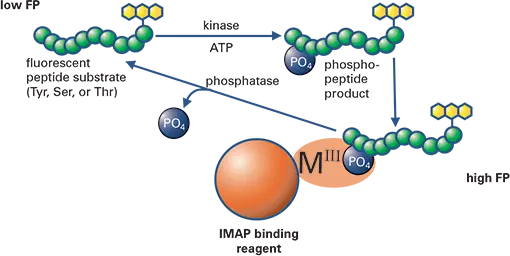
図1. IMAP FPアッセイの原理。IMAP FPキナーゼアッセイ原理図
SpectraMax® M5マルチモードマイクロプレートリーダーは、IMAPアッセイを用いたアッセイ開発やハイスループットスクリーニングに最適です。このモノクロメーターベースの装置では、追加のフィルターセットを購入することなく、選択した蛍光色素に最適な波長を選択することができます。このアプリケーションノートでは、SpectraMax M5 マイクロプレートリーダーと SoftMax® Pro ソフトウェアを使用して、緑色および赤色標識蛍光基質を用いた IMAP 蛍光偏光(FP)キナーゼアッセイと検量線を実行する方法についてご説明します。酵素希釈曲線は、T細胞シグナル伝達に重要な役割を果たすチロシンキナーゼであるLckと、ホスファチジルイノシトール3キナーゼシグナル伝達と細胞生存に関与するセリン-スレオニンキナーゼであるAkt1/ PKBαについて行いました。スタウロスポリンによるAkt1/PKBαの阻害もアッセイしました。FAMおよびTAMRA標識ペプチド基質の両方で高いZ' factor値が得られ、フィルターベースのAnalyst® HT Multi-Mode Microplate Readerで得られたデータと同等の結果が得られました。
材料
- プログレッシブ結合システム付きIMAP スクリーニング・エクスプレス キット(モレキュラーデバイス)
◦IMAPプログレッシブ結合試薬
◦IMAP Progressive Binding Buffer A(5X)
◦IMAP Progressive Binding Buffer B(5X)
◦IMAP Reaction Buffer(5X)
- Lckキナーゼ(Upstate)
◦FAM-p34cdc2由来ペプチド(モレキュラーデバイス)
◦TAMRA-p34cdc2 由来ペプチド(モレキュラーデバイス)
◦FAM-p34cdc2 由来リン酸化ペプチドキャリブレーター(モレキュラーデバイス)
- Akt1-PKBαキナーゼ (Upstate)
◦FAM-Crosstide(モレキュラーデバイス)
◦FAM-ホスホ-Crosstide(モレキュラーデバイス)
-
アデノシン5'三リン酸(ATP)、精製水中50 mMストック(Sigma)
-
DL-ジチオスレイトール(DTT)、精製水中100mMストック(Sigma)
-
スタウロスポリン(Biomol)
-
黒色384ウェルポリスチレンマイクロプレート(Corning)
-
SpectraMax M5マルチモードマイクロプレートリーダーとSoftMax Proソフトウェア(モレキュラーデバイス)
方法
キナーゼ反応
ステップ1: 1X IMAP Reaction Bufferに最終濃度1mMのDTTを加えてComplete Reaction Buffer(CRB)を調製します(100mM DTTストックの1:100希釈)。
ステップ2: FAMor TAMRA標識ペプチド基質の400 nM(4X)ストックをCRBで調製します(20 µMペプチドストックの1:50希釈)。
ステップ3: ATPの20 µM(4X)ストックをCRBで調製します(50 mM ATPストックの1:2500希釈)。
ステップ4:アッセイに必要な4倍濃度の酵素希釈系列を調製します。キナーゼ阻害アッセイには、一定濃度の酵素を使用し、スタウロスポリンまたは他のキナーゼ阻害剤の希釈系列を作成します。希釈にはCRBを使用します。
ステップ5:酵素アッセイウエルに以下のものをピペッティングして、キナーゼ反応をセットアップします:
- 5 µL CRB またはスタウロスポリン
- 5 µL 酵素(酵素バックグラウンドのないサンプルの場合は、5 µL CRB で代用します)
- 20-µM ATPストック 5 µL
- 400-nM ペプチドストック 5 mL
ステップ6:室温で1~1.5時間インキュベートします。
注:より詳細な手順については、IMAP 製品添付文書を参照してください。
キャリブレーションスタンダード
ステップ1: CRBに100-nMのペプチドストックを調製します(キナーゼアッセイと同じペプチド基質を使用します)。
ステップ2: CRBで100-nMリン酸化ペプチドストックを調製します(キナーゼアッセイで使用した基質のリン酸化バージョンを使用します)。
ステップ3:ペプチドとリン酸化ペプチドのストックを合わせて、表1に示すキャリブレーション用スタンダードとします。表1に示す量は、四重反復サンプルを調製するのに十分な量です。
|
キャリブレーションスタンダード (リン酸化率) |
µL ペプチド ストック |
µL リン酸化ペプチド
ストック |
|---|---|---|
| 0 | 100 | 0 |
| 12.5 | 87.5 | 12.5 |
| 25 | 75 | 25 |
| 50 | 50 | 50 |
| 100 | 0 | 100 |
表1. 検量線標準液の調製
ステップ 4: 20 µL CRB を含む緩衝液のみのバックグラウンド検体を含め、各キャリブレーショ ン標準物質を 20 µL ずつ 4 重ウエルに分注します。
結合反応
ステップ 1: 75% Progressive Binding Buffer A と 25% Progressive Binding Buffer B を混合し、Progressive Binding Buffer を調製します。
ステップ 2: Progressive Binding Buffer で Progressive Binding Reagent を 1:600 に希釈し、Binding Solution を調製します。
ステップ3:各アッセイウェルおよびキャリブレーションスタンダードウェル(バッファバックグラウンドサンプルを含む)に60μLの結合溶液をピペッティングします。
ステップ4:遮光し、室温で1時間インキュベートします。
SoftMax Proでテンプレートをセットアップし、SpectraMax M5マイクロプレートリーダーでプレートを読み取ります:
注:FAMおよびTAMRA標識基質で使用するIMAP FPプロトコルは、SoftMax Pro 5のBinding Assaysプロトコルフォルダーにあります。
ステップ1:使用する蛍光色素に特化したSoftMax Pro IMAP FPプロトコルを開きます。FAMまたはTAMRA以外のフルオロフォアを使用する場合は、波長設定を調整してください。SpectraMax M5マイクロプレートリーダーの設定を表2に示します。
| Read type | Endpoint | ||||||||||
| Read mode | Fluorescence polarization | ||||||||||
| Wavelengths |
|
||||||||||
| Sensitivity |
Readings: 100 PMT: high or medium |
||||||||||
| Automix | Off | ||||||||||
| AutoCalibrate | On | ||||||||||
| Assay plate type | 384-well Costar black | ||||||||||
| Wells to read | [ユーザーにより決定される] | ||||||||||
| Settling time | Delay: 100 ms | ||||||||||
| AutoRead | Off | ||||||||||
表 2. SpectraMax M5マイクロプレートリーダー用SoftMax ProのIMAP FP設定
ステップ2:バックグラウンド検体、キナーゼアッセイ検体、キャリブレーション標準物質を指定する実験テ ンプレートをセットアップします。キナーゼアッセイサンプルとキャリブレーション標準液は、SoftMax Proテンプレート内の8つの設定済み'Sample'グループに割り当てることができます。バッファーのみのコントロールを対応する'Background'グループに割り当てることで、ミリ偏光(mP)値を計算する前にバックグラウンド蛍光を自動的に差し引くことができます。
ステップ3:マイクロプレートをプレートリーダーキャリッジにセットし、Readをクリックします。
結果
SoftMax Pro IMAP FPプロトコルは、平行および垂直の平均値、mP、全強度、標準偏差、CVを自動的に計算します。各アッセイサンプルセットのグループテーブルでは、テンプレートで指定されたバックグラウンドサンプルをリファレンスとして、バックグラウンド減算mPが計算されます。リン酸化率は、適切な検量線から外挿することによっても算出できます。
図2は、FAM-p34cdc2由来ペプチドを用いて得られたLckキナーゼ希釈曲線を示します。このプレートを SpectraMax M5 マイクロプレートリーダーで読み取ると、酵素濃度範囲 0.04~3.5 unit/mL でデルタ mP は 303、Z' factorは 0.87 でした *2。 図 3 は、同じアッセイプレートを Analyst HT マルチモードリーダーで読み取ったものです。デルタmPは336で、Z' factorは0.95であった。どちらの装置でもEC50は0.5 unit/mLでした。
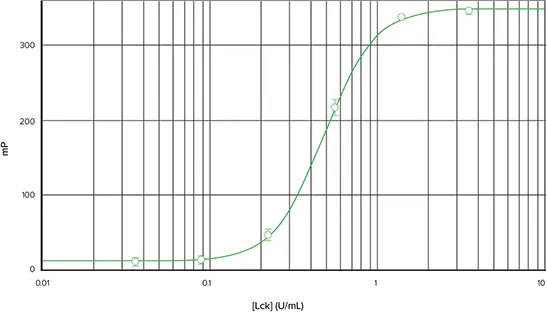
図2. SpectraMax M5マイクロプレートリーダーを用いたIMAP FPキナーゼアッセイ。SpectraMax M5で読み取った、0.04から3.5単位/mLの酵素にFAM-p34cdc2由来ペプチドを加えたLckキナーゼ希釈曲線です(4パラメータ曲線フィット)。エラーバーは標準偏差です。
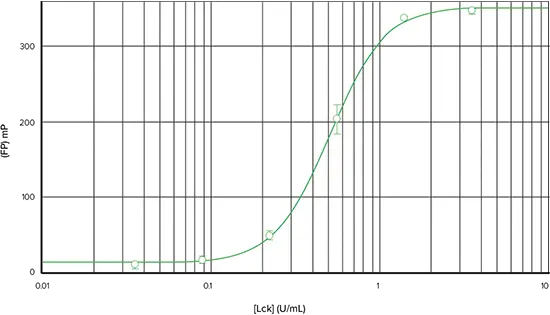
図3. Analyst HTを用いたIMAP FPキナーゼアッセイ。Analyst HTで読み取ったFAM-p34cdc2由来ペプチドによるLckキナーゼ希釈曲線です(0.04~3.5ユニット/mL酵素)(4パラメータ曲線フィット)。
非リン酸化ペプチド対照とリン酸化ペプチド対照を用いて別々の検量線を設定すれば、mPの値をリン酸化パーセンテージに変換することができます(図4)。まず新しいグラフを作成し('Graph#1'がソフトウェアによって割り当てられたデフォルトの名前です)、キャリブレーターサンプルグループのmP対リン酸化パーセント(濃度)をプロットします。このプロットのデフォルト名は 'Plot#1' です。検量線標準物質グループの表に、以下の式で新しい列を挿入します: InterpX('Plot#1@Graph#1@IMAP FP_FAM',AvgbkgsubmP)。'IMAP FP_FAM' は実験タイトルです。
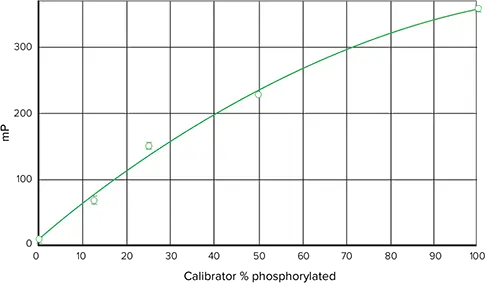
図4. FAM標識ペプチドを用いたIMAP。SpectraMax M5で読み取ったFAM-p34cdc2由来ペプチドとリン酸化ペプチドによる検量線標準曲線です(二次曲線フィット)。
赤色蛍光体を用いたIMAPキナーゼアッセイの性能を決定するために、TAMRA-p34cdc2-由来ペプチドを用いて別のLckキナーゼ希釈曲線を行いました(図5)。SpectraMax M5マイクロプレートリーダーで読み取ると、酵素濃度範囲0.04~3.5 unit/mLで、Z' factor 0.95、デルタmP258が得られました。図6では、同じアッセイプレートをAnalyst HTリーダーで読み取り、デルタmPは270、Z' factorは0.93でした。どちらの装置でもEC50は0.4 unit/mLで、FAM標識基質によるEC50に近い値でした。対応する検量線も設定されました(図7)。両装置でTAMRA標識ペプチド基質を用いた結果とFAM標識基質を用いた結果が類似していることから、ユーザーは、被験化合物から放出されるバックグラウンド蛍光を最小限に抑えるなど、赤色標識蛍光色素の利点を活用することができます。
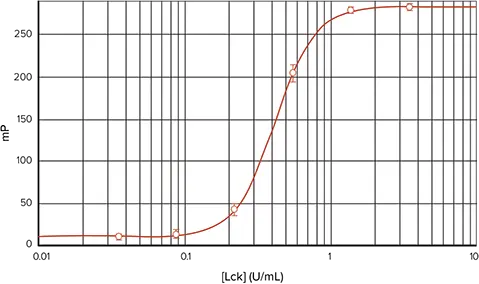
図5. SpectraMax M5マイクロプレートリーダーを用いたTAMRA標識ペプチドによるLckキナーゼアッセイ。SpectraMax M5で読み取ったTAMRA-p34cdc2由来ペプチドによるLckキナーゼ希釈曲線です(4-parameter curve fit)。
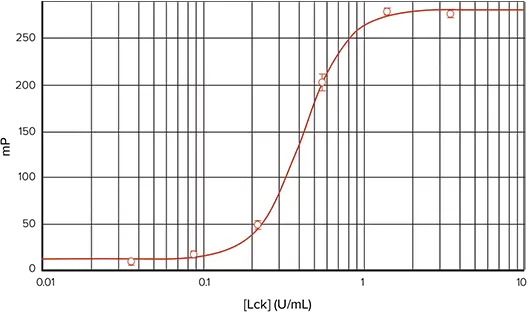
図6. Analyst HT を用いた TAMRA 標識ペプチドによる Lck キナーゼアッセイ。Analyst HTで読み取ったTAMRA-p34cdc2由来ペプチドによるLckキナーゼ希釈曲線です(4-parameter curve fit)。
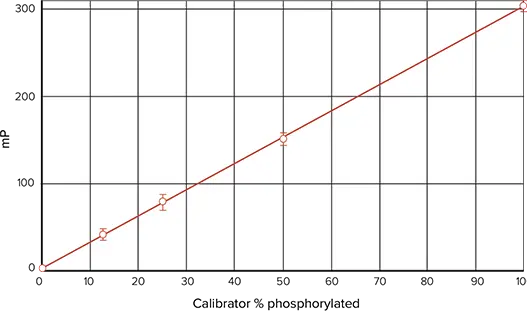
図7. TAMRA標識ペプチドを用いたIMAP。SpectraMax M5 で読み取りました TAMRA-p34cdc2 由来ペプチドとリン酸化ペプチドによる検量線標準曲線です(linear curve fit)。
別のキナーゼを用いた SpectraMax M5 マイクロプレートリーダーでの IMAP FP アッセイの性能を示すために、基質として FAM-Crosstide を用いて Akt1/PKBα の酵素希釈曲線を得ました。図8では、0.002~5.4 units/mLの酵素濃度範囲で、339のデルタmPと0.92のZ' factorが得られました。スタウロスポリンによるAkt1/PKBαの阻害を図9に示します;ここでのデルタmPは199で、Z' factorは0.93でした。このアッセイに対するスタウロスポリンのIC50の計算値は19.7nMでした。ここでは酵素希釈曲線よりもデルタmPが低かったが、これは阻害アッセイに約75%の最大リン酸化を与えるキナーゼ濃度が選ばれたためです。
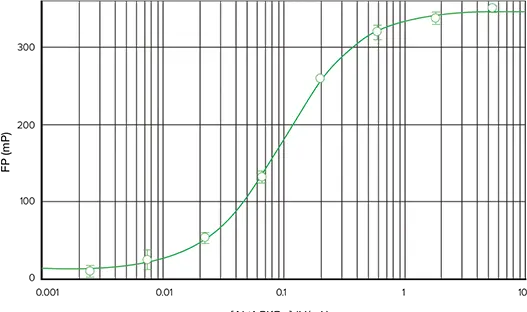
図8. SpectraMax M5マイクロプレートリーダーを用いたFAM標識ペプチドによるAkt/PBKaキナーゼアッセイ。SpectraMax M5で読み取ったFAM-Crosstide基質を用いたAkt1/PKBαキナーゼ希釈曲線です(4-parameter curve fit)。
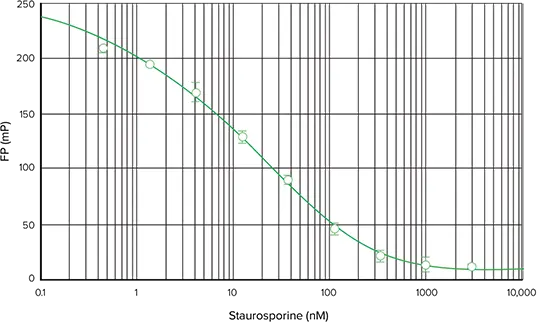
図9. SpectraMax M5マイクロプレートリーダーでのキナーゼ阻害。SpectraMax M5で読み取りました、スタウロスポリンとFAM-Crosstide基質を用いたAkt1/PKBαキナーゼ阻害曲線です。ここでは、SoftMax Pro 5で利用可能なfive-parameter curve fitを使用しました。
結論
細胞シグナル伝達や疾患におけるキナーゼの役割を理解し、治療薬となりうるキナーゼモジュレーターを同定しようとする研究者は、アッセイ開発とハイスループットスクリーニングの両方に適した非放射性のホモジニアスアッセイにますます注目しています。ホモジニアスFPアッセイは、キナーゼ活性化因子、阻害剤、基質を同定するためのHTSに適したフォーマットを提供します。SpectraMax M5 マイクロプレートリーダーで IMAP FP キナーゼアッセイを実行すると、正確で再現性のあるデータが得られ、Z' factor値も堅牢です。デュアルモノクロメーターシステムにより、追加のフィルターセットを購入することなく、幅広い蛍光標識ペプチド基質を用いてアッセイを最適化することができます。緑または赤で標識された基質を用いたIMAP FPキナーゼアッセイの結果は、フィルターベースのAnalystマイクロプレートリーダーで得られた結果と非常によく似ています。SoftMax Pro 5のIMAP FPプロトコールは、FPデータの取得と計算を行う便利な方法を提供し、あらゆる蛍光色素に簡単に適応できます。
参考文献
- IMAP Akt Assay Kit Product Insert(Molecular Devices)
- Zhang, J.H., Chung, T.D.Y., and Oldenburg, K.R. (1999). A simple statistical parameter for use in evaluation and validation of highthroughput screening assays. J Biomol Screen 4(2): 67-73.
PDF版(英語)