Application Note NanoBRETアッセイによる
p53-MDM2タンパク質相互作用の測定方法
- NanoLucのより明るいシグナルと広いスペクトル分離により、他のBRET技術よりも高感度を実現します
- 生細胞において、生理的レベルでタンパク質間相互作用を高感度に検出します
- SoftMax ProソフトウェアでNanoBRET比を計算し、結果を自動的にグラフ化します。
PDF版(英語)
はじめに
Joyce Itatani |アプリケーション・サイエンティスト|モレキュラーデバイス
Cathy Olsen, PhD|シニア・アプリケーション・サイエンティスト|モレキュラーデバイス
BRET(Bioluminescence Resonance Energy Transfer)は、発光ドナーと蛍光アクセプターの相互作用を利用して、タンパク質間またはタンパク質-リガンド間の相互作用を測定する技術です。ドナーとアクセプターが10 nm以内に近接すると、ドナーがアクセプターを励起し、アクセプターが蛍光を発します。関心のあるタンパク質の一方にドナーを、結合パートナーにアクセプターをタグ付けすることで、マイクロプレートリーダーを用いてドナーとアクセプターから発せられる光を検出し、タンパク質間相互作用を測定できます。
Promega社のNanoBRET™テクノロジーは、より明るい発光ドナー(NanoLucルシフェラーゼ)、最適化されたエネルギーアクセプター(HaloTag®-NCT)、およびドナーとアクセプターの波長間の広い分離を組み込むことで、BRET1やBRET2を含む従来のBRET技術を改良しています(図1)。これらの改良により、シグナルの増強、感度の向上、バックグラウンドの低減が実現し、生細胞内でのタンパク質間相互作用の検出が可能になります *1。
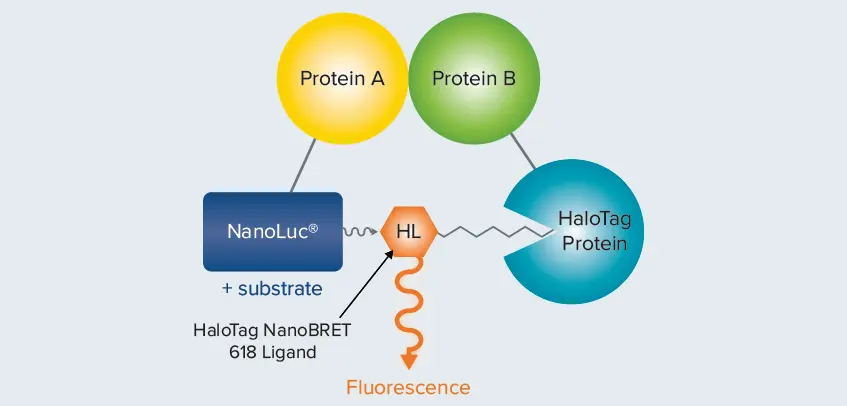
図1. NanoBRETアッセイ NanoLuc-Protein A融合体(エネルギードナー)が蛍光標識されたHaloTag-Protein B融合体(エネルギーアクセプター)と相互作用すると、ドナーとアクセプターが近接し、エネルギーが転移します。
NanoBRETシグナルの検出とデータ解析には、高感度な装置と高度なソフトウェアが必要です。SpectraMax® iD5 マルチモードマイクロプレートリーダーとSoftMax® Proソフトウェアを使用することで、最適化されたフィルターセットを用いてNanoBRETデータを取得し、カーブフィッティングを含む解析を適用できます。本稿では、SpectraMax iD5プレートリーダー上で、相互作用するタンパク質ペアp53とMDM2からなるNanoBRET™ PPIコントロールペアを用いたマイクロプレートリーダーの検証について説明します。p53経路の活性化剤であるnutlin-3を用いて、p53-MDM2相互作用を濃度依存的に阻害し、その結果をSoftMax Proソフトウェアで解析・グラフ化しました。
材料
- NanoBRET PPI コントロールペア (p53, MDM2; Promega)
- NanoBRET Nano-Glo ® 検出システム (Promega)
- ViaFect™ トランスフェクション試薬 (Promega)
- Nutlin-3(ミリポア-シグマ)
- Opti-MEM™ 還元血清培地、フェノールレッド不使用 (ThermoFisher)
- 293(HEK-293)セル (ATCC)
- イーグル最小必須培地(EMEM、Corning)
- BenchMark™ ウシ胎児血清(Gemini Bio-Products)
- ペニシリン-ストレプトマイシン(10,000 U/mL、ThermoFisher)
- 6ウェルクリアマイクロプレート(VWR)
- 96ウェル白色平底ポリスチレン・マイクロプレート(Corning)
- SpectraMax iD5 マルチモードマイクロプレートリーダー(モレキュラーデバイス)
⚪︎ドナーフィルター: ドナーフィルター:447 nm BW 60 nm(モレキュラーデバイス)
⚪︎アクセプターフィルター:610 nm LP(モレキュラーデバイス)
方法
HEK-293細胞を細胞培養培地(EMEM + 10% FBS + 1%ペニシリン/ストレプトマイシン)に懸濁し、400,000 cells/mLで6ウェルプレートに2 mL/well(800,000 cells/well)で播種しました。細胞は37°C、5% CO₂で4~6時間付着させ、その後、p53-HaloTag融合ベクターDNA 2 μgとNanoLuc-MDM2融合ベクターDNA 0.2 μgを、フェノールレッドを含まないOpti-MEM Reduced-Serum Medium 100 μL中で、ViaFectトランスフェクション試薬:DNA比3:1でトランスフェクトしました。細胞は37°C、5% CO₂で20~24時間インキュベートしました。
HEK-293細胞を1,000 rpmで5分間遠心し、培地を除去しました。細胞密度をOpti-MEM + 4% FBSで2.2 × 10⁵ cells/mLに調整し、2本の15 mLコニカルチューブに分注しました。一方にはHaloTag 618 Ligand 0.1 μMを添加し、もう一方にはリガンドを添加しませんでした。細胞は96ウェル白色マイクロプレートに2 × 10⁴ cells/wellで播種し、直ちにnutlin-3の1:3段階希釈液(各濃度n = 4)または0.5% DMSOで処理しました。細胞は37°C、5% CO₂で一晩インキュベートしました。
NanoBRET Nano-Glo基質の5X溶液をOpti-MEM + 4% FBSで調製し、各ウェルに25 μL添加しました。ドナー発光(447 nm)とアクセプター発光(610 nm)はSpectraMax iD5プレートリーダーで、表1に示す設定を用いて測定しました。NanoBRET比はSoftMax Proソフトウェアでアクセプターシグナルをドナーシグナルで割って算出し、比に1000を掛けてmilliBRET単位(mBU)を得ました。mBU値はリガンド非添加サンプルの平均mBUを差し引いてバックグラウンド補正しました。nutlin-3処理細胞の結果はSoftMax Proソフトウェア(バージョン7.0.3以上)で4パラメータカーブフィットを用いて解析・グラフ化しました。各濃度のnutlin-3でZ’因子を算出し、アッセイ性能を評価しました。
| Parameter | Setting |
|---|---|
| Read mode | LUM(ルミネッセンス) |
| Read type | Endpoint |
| Wavelengths | Specify wavelengths Use filter [make sure filters are installed] Lm1: 447 nm Lm2: 610 nm |
| Plate type | 96-well Costar |
| PMT and optics | Integration time: 1000 ms Read height: 0.67 mm* |
*Read heightは使用するマイクロプレートに合わせて最適化してください。表1. SpectraMax iD5プレートリーダーでのNanoBRET検出設定
結果
NanoBRET比(mBU)とnutlin-3濃度の関係をSoftMax Proソフトウェアで4パラメータカーブフィットを用いてグラフ化しました(図2)。nutlin-3のIC₅₀値は1.2 μMで、Promega社がNanoBRET PPIコントロールペアで示した結果と一致しました *2。
図2に示したカーブを生成するために使用したすべてのnutlin-3濃度で、Z’因子は少なくとも0.7を示しました。0.07 μM以下の濃度では、Z’因子は0.9でした。これらの値は、このNanoBRETアッセイの堅牢性と低い変動性を示しています。
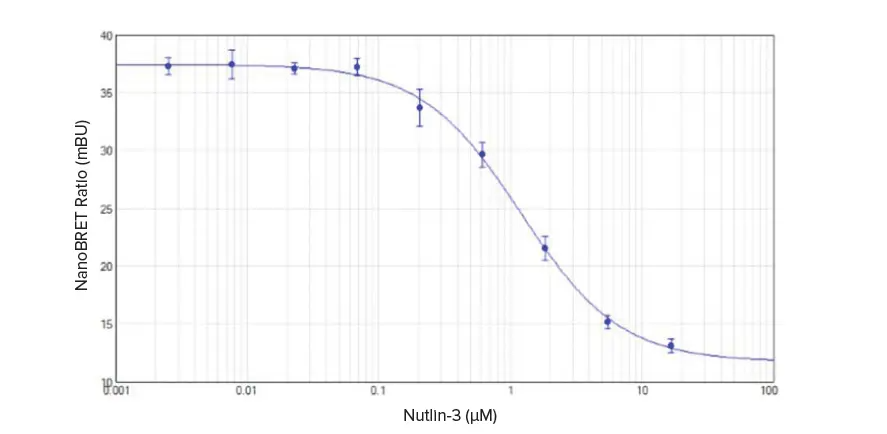
図2. nutlin-3によるp53-MDM2相互作用の阻害 SoftMax Proソフトウェアで生成した4パラメータカーブフィットのIC₅₀は1.2 μM(n = 4)でした。
結論
NanoBRETドナーおよびアクセプターシグナル検出用の最適フィルターを搭載したSpectraMax iD5プレートリーダーは、NanoBRET PPIコントロールペア(p53、MDM2)を用いた本アッセイで検証されました。nutlin-3は濃度依存的にp53-MDM2相互作用を阻害し、期待されるIC₅₀値1.2 μMを示しました。アッセイ性能と再現性を評価するために算出したZ’因子はすべての濃度で0.7~1の範囲にあり、SpectraMax iD5プレートリーダーの高感度とNanoBRETアッセイの堅牢性を確認しました。SoftMax ProソフトウェアはNanoBRET比の自動計算とデータのグラフ化を行い、解析を効率化しました。
参考文献
- Machleidt T, Woodroofe CC, Schwinn MK, Mendez J, Robers MB, Zimmerman K, Otto P, Daniels DL, Kirkland TA, and Wood KV. NanoBRET—A Novel BRET Platform for the Analysis of Protein-Protein Interactions. ACS Chem. Biol. 2015, 10, 1797–1804.
- Technical Manual: NanoBRET Protein: Protein Interaction System. Promega Corporation.
PDF版(英語)