Application Note SpectraMaxマイクロプレートリーダーを用いた
CyQUANT細胞増殖アッセイによる細胞増殖の測定
- 細胞増殖を迅速かつ簡便に定量化
- わずか50 cellsの高感度検出
- SoftMax Proソフトウェアのプロトコールで簡単データ解析
PDF版(英語)
はじめに
蛍光を用いた細胞増殖の定量により、細胞増殖に対する薬剤やその他の実験的処置の効果を容易にモニターすることができます。Life Technologies 社の CyQUANT Cell Proliferation Assay Kit は、蛍光マイクロプレートリーダーを用いた高感度、迅速かつ簡便な細胞増殖定量法です。CyQUANT GR色素は細胞核酸に結合し、標準曲線から細胞数を算出することができます。DNA 対 RNA 比は細胞周期の過程で変化する可能性があるため、CyQUANT キットを使用すると、RNase 消化細胞溶解液と核酸標準曲線を用いて細胞数を測定することができます。
このアプリケーションノートでは、CyQUANTキットをモレキュラーデバイスのSpectraMax®マイクロプレートリーダーおよびSoftMax® Proソフトウェアと組み合わせて使用する方法についてご説明します。2つの方法について詳述しますが、一つ目は、細胞ベースの標準曲線を用いて細胞増殖を定量する方法で、もう一つは、RNase処理した細胞サンプルとDNA標準曲線を用いて細胞増殖を定量する方法です。
材料
- CyQUANT Cell Proliferation Assay Kit(Life Technologies): コンポーネントA、400XのCyQUANT GR色素、コンポーネントB、20Xの細胞溶解バッファー、およびコンポーネントC、λ DNAスタンダード、100 µg/mL溶液100 µL。
注意:Lysis Buffer と CyQUANT GR は希釈後数時間以内に使用すること。CyQUANT GR 溶液は必ず遮光する。 - セル 本アプリケーションノートのデータ作成にはCHO-K1細胞を使用した(ATCC)
- DNA 標準曲線を使用する場合は EDTA(Fisher Chemicals)
- DNA 標準曲線を使用する場合は NaCl(Fisher Chemicals)
- DNase フリーの RNase A または RNase カクテル(Ambion)
- 黒色96ウェルクリアボトムマイクロプレート(Corning)
- 蛍光検出モード付きSpectraMaxマイクロプレートリーダー(モレキュラーデバイス)
細胞の調製
接着細胞または非接着細胞は、CyQUANT アッセイ用に、アッセイするマイクロプレート内で直接増殖させ凍結させるか、培養容器内で増殖させた後、アッセイ用にマイクロプレートに移す細胞のペレットサンプルを凍結させることにより調製しました。詳細は、Life Technologies 社の CyQUANT Cell Proliferation Assay 製品情報シート MP-7026 をご参照ください。
細胞ベース標準曲線を用いたCyQUANTアッセイの実施方法
増殖アッセイする細胞を準備
ステップ 1: 培養プレートからトリプシンまたは EDTA 溶液を用いて接着細胞を剥離し、培地を用いて細胞懸濁液を 96 ウェルマイクロプレートに希釈し、表 1 のテンプレートセットアップに示す細胞濃度(cells/well)にします。細胞を含まない対照ウェルも用意します。
| Well | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| A | 50,000 | 25,000 | 12,500 | 6,250 | 3,125 | 1562 | 391 | 195 | 98 | 49 | 24 | no cells |
| B | 50,000 | 25,000 | 12,500 | 6,250 | 3,125 | 1562 | 391 | 195 | 98 | 49 | 24 | no cells |
| C | 50,000 | 25,000 | 12,500 | 6,250 | 3,125 | 1562 | 391 | 195 | 98 | 49 | 24 | no cells |
| D | 50,000 | 25,000 | 12,500 | 6,250 | 3,125 | 1562 | 391 | 195 | 98 | 49 | 24 | no cells |
表1. マイクロプレートに調製した細胞濃度(細胞数)
ステップ 2: マイクロプレートを 37℃で所望の時間インキュベートし、その後増殖を測定。インキュベーション時間は、実施する増殖アッセイのタイプ、使用する細胞タイプ、実験デザインによって異なります。
ステップ 3: ウェルから培地を静かに吸引します。ウェルはリン酸緩衝生理食塩水(PBS)で穏やかに洗浄してもよいですが、これは必須ではなく、細胞が外れる可能性のある高密度の培養には推奨されません。
ステップ4:マイクロプレート内の細胞を、アッセイ準備が整うまで-70℃で凍結(最低1時間、最長4週間)。この凍結ステップにより、細胞サンプルの完全な溶解が保証されます。
標準曲線用の細胞の準備
注意:標準曲線用の細胞は、実験で使用したものと同じ種類を使用するのが最善です。
ステップ1:トリプシンまたはEDTA溶液を用いて培養プレートから接着細胞を剥離し、培養メデュームに濃縮細胞懸濁液を調製する。細胞は105~106個/mL程度の密度にします。
ステップ2:細胞懸濁液1.0 mLを約200 x g(微量遠心機で約1500 rpm)で5分間遠心します。上清を捨て、細胞ペレットを-70℃で凍結。この凍結工程により、細胞が完全に溶解されます。
アッセイ試薬の調製
ステップ 1 : 濃縮細胞溶解バッファー原液を蒸留水で1:20に希釈し、1X細胞溶解バッファーとします。
ステップ 2 : 濃縮 CyQUANT GR 原液を 1:400 に希釈し、1X 細胞溶解バッファーを調製。この溶液はガラスではなくプラスチック容器に調製します(CyQUANT 製品添付文書参照)。
セルベース標準曲線および未知検体の調製
ステップ 1 : 凍結しておいたセルペレットを室温で数分間解凍。CyQUANT GR dye/cell-lysis buffer 1.0 mL を加え、短時間ボルテックスして細胞を再懸濁します。ペレットに106個の細胞が含まれていたと仮定すると、得られた細胞溶解液には106個/mL相当の細胞が含まれていることになります。
ステップ2:マイクロプレートのウェルに希釈系列を作り、50,000 cells/wellから開始し、24細胞/ウェルまで1:2希釈系列を作ります。CyQUANT GR dye/cell-lysis buffer を使用し、目的の細胞数が 200 µl の容量になるように希釈。各標準を4レプリケート調製し、コントロールとして細胞無しのレプリケートを1セット含みます(マイクロプレートに余裕を持たせるため、標準細胞の1つ(例:781 cells/well)を省略することができます)。
ステップ3:マイクロプレートを遮光し、室温で2~5分間インキュベート。
設定とソフトウェアのセットアップ
ステップ1:マイクロプレートリーダーの電源を入れます。SoftMax Proソフトウェアを起動し、Protocol LibraryのCell Growth & ViabilityフォルダーにあるCyQUANT Fluorescenceプロトコルを開きます。
ステップ2:表2に示設定を使用します。これらの設定は、設定済みプロトコールですでに選択されています。
| 設定 | |
|---|---|
| リードモード | FL |
| リードタイプ | Endpoint |
| 波長 | 励起485 nm、発光538 nm、カットオフ515 nm |
| プレーティングタイプ | 96ウェル標準クリアボトム(デフォルト)、96ウェル標準不透明 |
| リードエリア | [ユーザーによって決定] |
| PMTと光学系 | PMTゲイン: 自動、読み取りごとのフラッシュ数:10 |
| シェイク | 初読前:5秒 |
| その他の設定 | [ユーザーにより決定、プレートリーダー特異性] |
表2. マイクロプレートリーダーの設定 SpectraMax M5 プレートリーダーの設定を示します。蛍光検出モードを持つ他のモレキュラーデバイス製プレートリーダーについても同様の設定を行ってください。
ステップ3:プロトコールの細胞標準セクションにある Template Editor を使用して、マイクロプレート内の標準品(各希釈の細胞番号)、ブランク(細胞溶解液なし)、未知物質の位置を示すアッセイのテンプレートを作成。標準品と未知検体のテンプレートの例を図1に示します。CtrlキーとShiftキーを同時に押したときに表示されるテンプレートセットアップウィンドウで、各ウェルに割り当てられた値が表示されていることに注意してください。未知検体の割り当て名(Un01-Un12)はこのビューには表示されません。
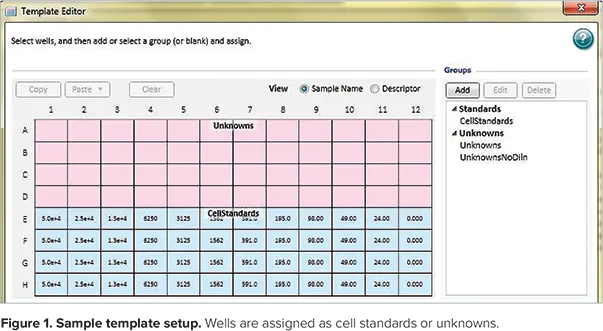
図1. テンプレートのセットアップ例。ウェルは細胞標準物質または未知物質として割り当てられます。
ステップ4:マイクロプレートをマイクロプレートリーダーにセットします。ご使用のマイクロプレートリーダーでトップリード用のアダプターが必要な場合(SpectraMax M5など)は、プレートキャリッジにアダプターをセットします。
ステップ5:ソフトウェアのReadボタンをクリックします。設定がプレートを読み取り、SoftMax ProソフトウェアのPlateセクションに相対蛍光単位(RFU)が表示されます。
データの分析
ステップ1: マイクロプレートの読み取りが完了すると、テンプレート設定時に自動的に作成されたGroup tableでデータが解析されます。
ステップ2: Template EditorのStandardsグループにウェルが割り当てられていれば、ソフトウェアによって標準曲線が自動的にプロットされます。グラフセクションのドロップダウンカーブフィットメニューから適切なカーブフィット処理を選択します。このアプリケーションノートで標準曲線をプロットする際、log-logカーブフィットを使用しました(図2)。二次曲線フィットも適しているかもしれません。
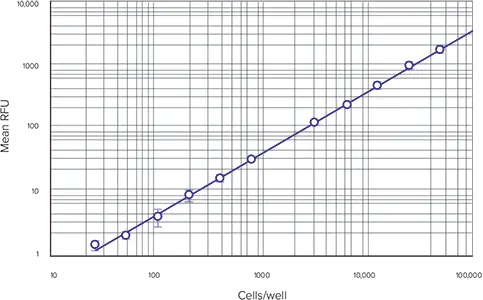
図2. 細胞ベースの標準曲線 細胞密度(1ウェルあたり25~50,000細胞)を測定。
ステップ3:実験細胞サンプル(未知)のウェルあたりの細胞数を決定します。SoftMax Pro CyQUANTアッセイプロトコールでは、細胞ベース標準曲線からこれらの値を算出し、UnknownsグループセクションのConcentration列に記載します。
細胞ベース標準曲線の結果
上記のように設定した細胞ベースの標準曲線を図 2 に示します。標準曲線をアッセイの全ダイナミックレンジにわたってグラフ化する際、log-logカーブフィットを使用。結果はSpectraMax M5マイクロプレートリーダーによるものですが、蛍光検出モードを備えた他のモレキュラーデバイス マイクロプレートリーダーでも同様の結果が得られます(データは示していません)。
表3は、表1のスキームに従ってプレーティングし、2日間培養で増殖させたCHO-K1細胞の細胞増殖アッセイ結果のサンプルです。2日目の細胞/ウェルは、細胞ベースの標準曲線を用いてSoftMax Proで算出しました。
| サンプル数 | プレーティングした細胞数 0日目 | 平均細胞数 2日目 | 標準偏差 | CV(%) |
|---|---|---|---|---|
| Un01 | 50,000 | 58,861 | 6255 | 10.6 |
| Un02 | 25,000 | 56,822 | 8055 | 14.2 |
| Un03 | 12,500 | 54,860 | 5203 | 9.5 |
| Un04 | 6250 | 47,854 | 7548 | 15.8 |
| Un05 | 3125 | 32,746 | 6257 | 19.1 |
| Un06 | 1562 | 23,036 | 4533 | 19.7 |
| Un07 | 391 | 9262 | 2884 | 31.1 |
| Un08 | 195 | 5198 | 1873 | 36.0 |
| Un09 | 98 | 3856 | 714 | 18.5 |
| Un10 | 49 | 2690 | 814 | 30.3 |
| Un11 | 24 | 2208 | 179 | 38.1 |
表3. 未知グループデータの例 細胞の平均濃度(cells/well)はプレーティングの2日後に算出しました。
DNAベースの標準曲線とRNase処理細胞を用いたCyQUANTアッセイの実施方法
細胞増殖は、DNA標準曲線を用いてDNA含量に基づいてアッセイすることもできます。標準曲線は、RNA が蛍光シグナルに寄与しないように細胞溶解液を DNase フリー RNase で前処理すれば、細胞 DNA を定量することができます。このアプリケーションノートでは、DNA スタンダードカーブの設定方法と RNase 処理した細胞サンプルについて説明します。
アッセイ試薬の準備
ステップ 1: 濃縮細胞溶解バッファー原液を蒸留水で1:20に希釈し(1ウェルあたり200 µL調製)、1X溶解バッファー溶液を調製。最終濃度が1mM EDTAと180mM NaClのものと、EDTAやNaClを含まないものの2種類の1X溶解バッファーを用意します。
ステップ 2: 濃縮 CyQUANT GR 原液をそれぞれ 1:400 または 1:200 に希釈し、CyQUANT GR 色素/細胞溶解バッファー の 1X および 2X 作業溶液(各ウェルあたり 100 µL)を作製。この溶液は、Life Technologies 社が推奨するように、ガラス製ではなくプラスチック製の容器で調製します。
プラスチックチューブでDNAスタンダードを調製
ステップ 1: キットの 100 µg/mL DNA スタンダードを 1X CyQUANT GR dye/cell lysis buffer で希釈し、1 µg/mL のストック溶液を調製。
ステップ 2: 1 µg/mL 原液をさらに希釈し、表 4 に示す希釈系列を作成します。
| チューブ番号 | 1x CQ GR/Lysis Buffer(µL) | 1x CQ GR/Lysis Buffer中の1µg/mL DNA(µL) | 最終DNA濃度(ng/mL) |
|---|---|---|---|
| 1 | 1000 | 0 | 0 |
| 2 | 990 | 10 | 10 |
| 3 | 950 | 50 | 50 |
| 4 | 900 | 100 | 100 |
| 5 | 800 | 200 | 200 |
| 6 | 600 | 400 | 400 |
| 7 | 400 | 600 | 600 |
| 8 | 200 | 800 | 800 |
| 9 | 0 | 1000 | 1000 |
表4. DNAスタンダードの調製 DNAスタンダードを10~1000ng/mL、ブランクになるように指示通りに希釈しました。
注:CyQUANTキットにはλバクテリオファージDNAスタンダードが含まれています。このアプリケーションノートに示されたデータは、連続希釈で調製したこの標準物質を用いて作成されたものです。実施するアッセイによっては、キットの DNA スタンダード、または製品情報シート MP-7026 の指示に従って調製したサンプルに適した別の DNA を使用することをお勧めします。
細胞の準備
Life Technologies 社の CyQUANT 製品情報シートから、細胞の種類(接着細胞または非接着細胞)および培養条件(マイクロプレートまたは培養容器)に応じて、適切な 培養・凍結手順書を選択。細胞を-70℃で凍結します。このアプリケーションノートでは、接着細胞をトリプシン処理して培養容器から剥離し、ペレット化して凍結。アッセイ性能を示すために、既知数の細胞を封じ込めたサンプルを使用しました。
細胞サンプル、DNAスタンダードカーブ、ブランクの準備
ステップ1:事前に準備した凍結細胞ペレット(またはマイクロプレート)を-70℃保存から取り出します。凍結細胞を室温で数分間解凍します。
ステップ2:180mMのNaClと1mMのEDTAを封じ込めた100μLの1X細胞溶解バッファー を各ウェルと細胞のない4ウェル(ブランク)に加え、細胞をRNaseで前処理。ペレット化した細胞を使用する場合は、各サンプルを100µLに再懸濁し、マイクロプレートのウェルに移します。細胞を封じ込めた各ウエルと、細胞を封じ込めなかったコントロールのウエルに、4µLのRNase(各ウエルに2ユニット、下記注参照)を加えます。室温で1時間インキュベートします。
注意:DNaseフリーのRNaseは、サンプル処理に必要な全ユニット数が10 µL以下になるような濃度で使用。例えば、Ambion 社の RNase Cocktail は 500 U/mL の活性を持つので、1 ウェルあたり 4 µL で 2 ユニットとなります。
ステップ 3: 2X CyQUANT GR dye/cell-lysis buffer を、細胞のないコントロールウェルも含め、各マイクロプレートウェルに 100 µL ずつ加えます。
ステップ 4: DNA 標準曲線を作成するために、あらかじめ調製しておいた DNA 連続希釈液を、テンプレートエディターで定義したマイクロプレートの適切なウェルに移します。プレートには2つのコントロールを封入。細胞サンプルと同じ手順で調製した、細胞を含まない(DNAを含まない)消化コントロールと、200µLの1X CyQUANT GR色素/細胞溶解緩衝液のみを含む(DNAおよびRNaseを含まない)標準曲線コントロール/プレートブランクです。
ステップ5:サンプルを2~5分間インキュベートします。
設定とソフトウェアのセットアップ
ステップ1:マイクロプレートリーダーの電源を入れます。SoftMax Proソフトウェアを起動し、Protocol LibraryのCell Growth & ViabilityフォルダーにあるCyQUANT Fluorescenceプロトコルを開きます。
ステップ2:表2に示す設定を使用します。これらの設定は、設定済みプロトコールですでに選択されています。
ステップ3:プロトコールのDNAスタンダードセクションにあるTemplate Editorを使用して、マイクロプレート上のスタンダード、未知細胞(RNaseで処理した細胞)、プレートブランクの位置を示すアッセイのテンプレートを作成します。
プレートの読み取りとデータ解析
ステップ1:マイクロプレートをリーダーにセット。
ステップ2:プレートを読み取ります。
ステップ 3 : マイクロプレートの読み取り後、RFUがPlateセクションに表示されます。データは、テンプレートのセットアップ中に作成したグループテーブルで分析されます。
ステップ4: Template Editorでウェルが適切なグループに割り当てられていれば、DNAスタンダードカーブが自動的にプロットされます。
ステップ5:GraphセクションのCurve Fitメニューから適切なカーブフィット処理を選択します。この実験ではlog-logカーブフィットを使用しました。
ステップ6: DNA標準曲線から細胞DNA濃度が自動的に補間され、Unknownsグループの表に表示されます。
DNAスタンダード曲線の結果
典型的な DNA ベースの標準曲線を図 3 に示します。これらのデータの作成にはSpectraMax M5マイクロプレートリーダーを使用しましたが、蛍光検出モードを備えた他のモレキュラーデバイス製プレートリーダーでも同様の結果が得られます(データは示していません)。DNAスタンダードカーブからRNas処理細胞の細胞DNA濃度を算出した例を表5に示します。このアプリケーションノートでは、未知細胞として連続細胞希釈液を用いました。実際には、50~50,000 cells/well(標準曲線の直線範囲内)であれば、どのような細胞数でも検出可能であり、この方法を用いて細胞DNA値を求めることができます。
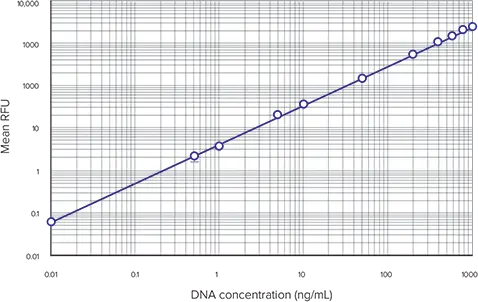
図3. DNAスタンダードカーブ CyQUANTアッセイキットを用いて得られたDNAスタンダードカーブ。
| cells/well | 平均RFU | 平均細胞DNA(ng) | 標準偏差 細胞DNA | CV(%) |
|---|---|---|---|---|
| 50,000 | 554.8 | 40.29 | 1.20 | 3 |
| 25,000 | 375.6 | 26.40 | 0.40 | 1.6 |
| 12,500 | 195.8 | 13.04 | 0.46 | 3.5 |
| 6,250 | 106.1 | 6.71 | 0.34 | 5 |
| 3,125 | 58.5 | 3.52 | 0.24 | 6.8 |
| 1,563 | 30.7 | 1.75 | 0.12 | 6.7 |
| 781 | 17.0 | 0.93 | 0.16 | 17.4 |
| 391 | 7.1 | 0.36 | 0.08 | 21.8 |
| 195 | 6.2 | 6.2 | 0.05 | 14.9 |
| 98 | 3.0 | 0.14 | 0.04 | 27.8 |
| 49 | 2.7 | 0.13 | 0.03 | 20.8 |
表5. サンプルグループの表: SoftMax® ProソフトウェアがDNAスタンダード曲線を用いて算出した細胞DNA濃度 グループ表のこのビューでは、リプリケートは表示されていません。
図4は、cells/wellに対する各サンプルセットの平均細胞DNAを示します。これらの条件下でのアッセイの直線範囲は、製品情報シート MP 7026 に基づく予想通り、50~50,000 cellsです。RNase 処理した細胞サンプルと未処理の細胞サンプルの RFU の比較を図 5 に示し、RNase 処理により細胞溶解液中の蛍光量が減少することを示しています。
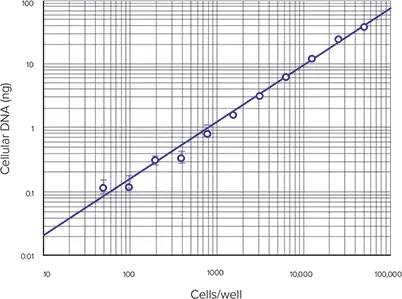
図4. 細胞DNA対ウェルあたりの細胞数 細胞DNA濃度とウェルあたりのRNase処理細胞数。
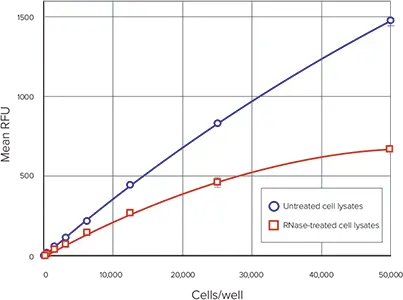
図5. RNase処理細胞と未処理細胞の比較 RNas処理細胞と未処理細胞のRFU vs cells/well。青丸は未処理細胞溶解液、赤四角はRNase処理細胞溶解液。
結論
CyQUANT Cell Proliferation Assay は、細胞数または細胞 DNA 含量を定量するための迅速で高感度な蛍光法です。アッセイ結果は、SpectraMax M5または蛍光検出モードを備えた他のモレキュラーデバイス プレートリーダーでも同様です。細胞ベースの標準曲線を用い、5分間のインキュベーションを行ったところ、CyQUANT製品情報シートに記載されているのと同様の検出限界とダイナミックレンジが確認されました(サンプルあたり50~50,000 cells)。キットに付属のλ DNAスタンダードで作成したDNA標準曲線を用いると、わずか50 cells中のDNA量を算出することができました。SoftMax Proソフトウェアの解析機能と、あらかじめ設定されたCyQUANTプロトコールを使用することで、データの計算と報告を簡便に行うことができます。
参考文献
Life Technologies 製品情報シート MP 7026 1/12/01: CyQUANT Cell Proliferation Assay Kit.
PDF版(英語)