Application Note SpectraMax PLUS マイクロプレートリーダーでの
タンパク質溶液の測定に適用される PathCheck
PDF版(英語)
はじめに
タンパク質の濃度は、280 nmの吸光度を測定し、タンパク質の消光係数を用いて濃度を計算することで推定するのが一般的です。消光係数は通常、1cmの光路長を固定した標準キュベットを用いて得られますので、この方法は実行可能です。消光に基づくタンパク質アッセイ法はマイクロプレートでも可能ですが、マイクロプレートウェル内のサンプルの光路長が変化するのを補正するのが面倒なため、あまり利用されてこなかったです。モレキュラーデバイスのSpectraMax PLUS マイクロプレートリーダーの登場は、マイクロプレートフォーマットで行われた測光値を自動的に正規化し、標準的な1cm光路長キュベット内の対応する溶液で得られた値と同じにすることができる初めての試みです。以下のアプリケーションノートでは、光路長補正機能 "PathCheck "の使用方法、キュベットとマイクロプレートで得られた吸光度結果の比較、カラムクロマトグラフィーのフラクションにPathCheckを適用し、溶出プロファイルを表示した例を紹介しています。
材料
光路長補正の原理の詳細については、MAXline アプリケーションノート#25「SpectraMax PLUS マイクロプレートリーダーにおける光路長 1 cm に自動的に正規化された光学密度測定」をご参照ください。
-
SpectraMax PLUS マイクロプレートリーダー
-
UV透過マイクロプレート:
◦SPECTRAplate ® Quartz、モレキュラーデバイス
◦SPECTRAplate Quartz ハーフエリアマイクロプレート、モレキュラーデバイス
◦Quartz microtest 8ウェルストリップ、Hellma;8ウェルストリップ用フレーム、E & K Scientific Productsと併用。注:Evergreen Scientific社から販売されている8ウェルストリップ用フレームは 正しく適合しません。
◦使い捨てUVプレート、Corning Costar Corp
◦UV-MAX(ポリフィルトロニクス) -
マイクロプレート用のピペッターとチップまたはトランスファーピペット。
-
サンプル(各100~300 µL)
-
石英キュベット
読み取り前測定のセットアップ
ステップ1 SoftMax Pro®を起動し、Plateセクションを開くか、必要に応じて新しいPlateセクションを作成します。
図1に示すように、[Instrument Settings]ダイアログボックスを設定します。装置設定ダイアログボックスで、280 nmで終点読み取りを実行するように選択します。PathCheckを選択し(プレリードプレートは自動的にチェックされます)、Cuvette Referenceを選択します。(別の方法として、工場出荷時にインストールされているWater Constantをショートカットとして使用することもできますが、精度が落ちる可能性があります)。
ステップ 2 装置設定ダイアログボックスを図 1 のように設定します。装置設定ダイアログボックスで、280 nmで終点読み取りを実行するように選択します。PathCheckを選択し(プレリードプレートは自動的にチェックされます)、Cuvette Referenceを選択します。(別の方法として、工場出荷時にインストールされているWater Constantをショートカットとして使用することもできますが、精度が落ちる可能性があります)。
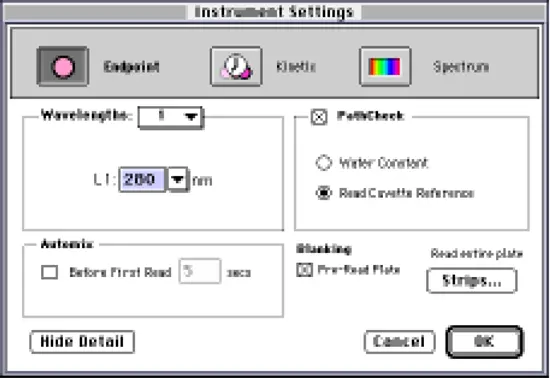
図1:PathCheckで280 nmの吸光度を測定するために設定された装置の設定ダイアログボックス
ステップ 3 Template Editor を使用して、マイクロプレート上の標準品、ブランク、未知検体の 配置を示すテンプレートを作成します。
ステップ4 図2に示すように、Reductionダイアログボックスを設定します。波長の組み合わせを L1 に、データモードを吸光度に設定します。「Use preread plate」と「Apply Pathcheck」が選択されていることを確認します。
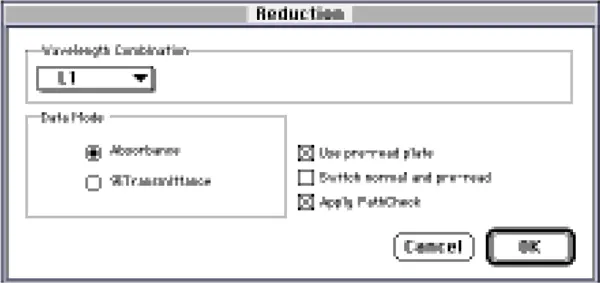
図2:PathCheckを適用するために設定されたReductionダイアログボックス
測定方法
プレリード測定
プレリードの目的は、プレートのバックグラウンド吸光度を差し引くことです。一方、ブランク(プレート、サンプル、グループ)はテンプレートで指定され、パス長補正後に差し引かれます。場合によっては、保存されているプレリード値を使用することもできます(下記参照)。
ステップ1 清潔なマイクロプレートを選び、蒸留水を目的のサンプルとほぼ同量のウェルにピペッ トします。正確なピペッティングは必要ありません。
ステップ 2 プレートを SpectraMax PLUS のドロワーに入れ、SoftMax Pro のツールバーにある Read ボタンをクリックして、プレリード測定を行います。
経路長補正吸光度測定の実行
注:事前にプログラムされたWater Constantを使用する場合は、ステップ1を省略できます。
ステップ 1 サンプルバッファーまたは蒸留水を入れた清潔な石英キュベットを SpectraMax PLUS のキュベットポートにセットします。
ステップ 2 サンプルとブランクの分注量をマイクロプレートの指定ウェルに移します。正確なピペッティングは必要ありませんが、最良の経路長補正結果を得るためには100 µLから300 µLの間の容量が必要です。
ステップ 3 プレートを SpectraMax PLUS のドロワーに入れ、SoftMax Pro のツールバーの Read ボタンをクリックして吸光度を測定します。(プレートの読み取りと同時にキュベットも近赤外光で読み取り、その吸光度値を PathCheck の計算に使用します)。
蒸発誤差を避けるため、マイクロプレートにサンプルを入れてから数分以内に測定を行ってください。読み取りを遅らせなければならない場合は、粘着シールでプレートを覆います。プレートを読み取る直前にシールを剥がしてください。
読み取り前データの保存と再利用
石英プレートは非常に均一であるため、ウェルごとにプレートのバックグラウンドを差し引く必要はありません。その代わりに、1枚のプレートを事前に読み取り、その事前読み取りデータをSoftMax Proファイルに保存して再利用することができます。
Costar UVプレートを使用する場合、必要な精度に応じて、保存されたプレリード値を使用することも、使用しないこともできます。280nmにおいて、水を含むCostarウェルの平均吸光度は約0.050+0.002 ODです。したがって、生の吸光度値が1 ODのサンプルは、プレートのバックグラウンド変動による潜在的な誤差が1%未満となります。しかし、ウェルの吸光度が低ければ低いほど、プレートのばらつきによる潜在的な誤差の最大値は大きくなります。
結果
例 1: タンパク質標準曲線を用いたPathCheckの有用性の実証
装置の性能を説明するために、タンパク質の「標準曲線」を作成しました。ウシ血清アルブミン(BSA)をpH7.4のリン酸緩衝生理食塩水(PBS)に溶解し、フィルター滅菌後、6、5、4、3、2、1、0.5、0.25 mg/mLに希釈しました。マイクロプレートで測定した吸光度を、同じ溶液を1cmキュベットで測定した吸光度と比較しました。
SoftMax Pro を起動し、「Plate Standard(プレート標準)」と題するグループを作成し、プレートセクションの Template Editor で「Standards(標準)」形式を指定しました。図 3 に示すように、標準物質をウェルに割り当てました。「スタンダード 01 "はタンパク質無添加の PBS(0mg/mL)でした。キュベットセットの Template Editor で、グループ名を「キュベット標準」とした以外は同じテンプレートを設定しました。
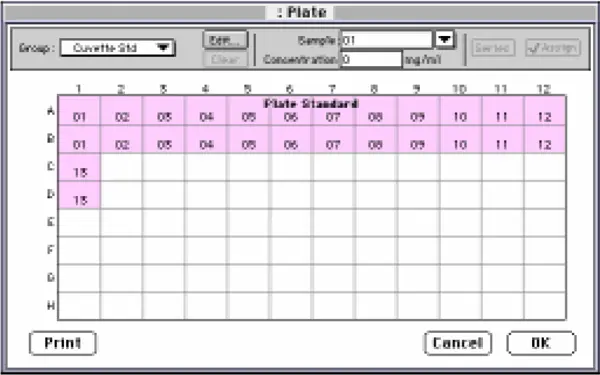
図3:Plateセクションのテンプレート・エディター
Plate セクションの装置設定とデータ削減は、上記の図 1 および図 2 に示すように設定しました。CuvetteSet セクションは 280 nm で終点を読み取るように設定しました。
PathCheck用のCuvetteリファレンスとして、PBSを入れたCuvetteをCuvetteポートにセットしました。プレリード測定は、Costar UVプレートの各ウェルに200μLの水を入れて行い、その後プレートを空にしてブロットして乾燥させました。タンパク質の「標準」溶液のアリコートを、使い捨ての移し替えピペットを使って指定されたウェルに移し替えました。PathCheck機能の有用性を示すため、各ウェルの容量は意図的に変化させました。プレートをマイクロプレートリーダーの引き出しに入れ、読み取りました。
CuvetteSet では、Cuvetteポート内の PBS 入りCuvetteを基準として読み取りを行いました。次に、各タンパク質「標準」溶液を 1 つずつCuvetteで読み取りました。
Plateセクションに関連するグループ表を図 4 に示します。
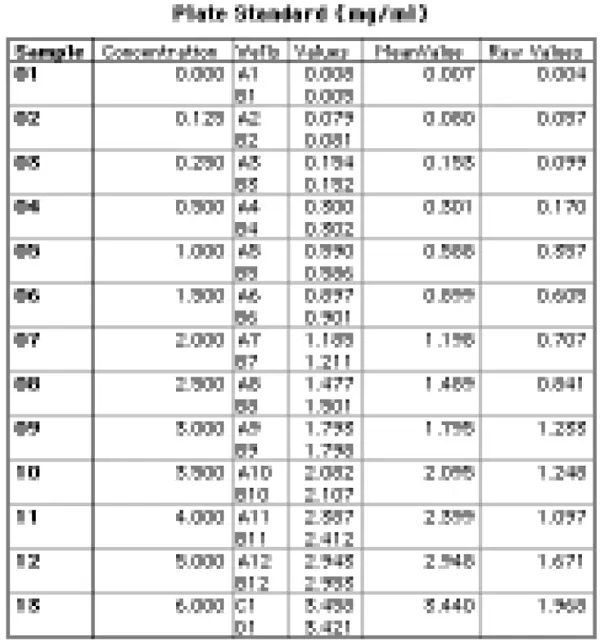
図4:Plateセクションのグループ・テーブル
グループテーブルの右端の列を除くすべての列は、テンプレートで「Plate Standard」グループを作成したときに、SoftMax Proが自動的に作成したものです。「Values」列の数値は、光路長1.0cmに補正した吸光度値です。右端の "Raw Values "列の数値は、光路長補正前の生の吸光度値(プレリード減算値)です。これらの値は、その列の計算ダイアログボックス(図5)に示されているカスタム計算式を使って逆計算することで得られました。基本的に、Raw値は補正後の値に対応する光路長を掛け合わせ、レプリケートの平均を取ることで算出しました。
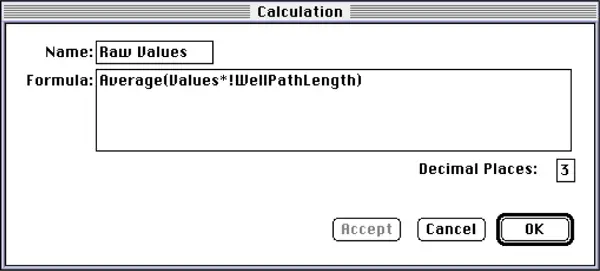
図5:"Raw Values "列の計算ダイアログボックス
グループ・テーブル(「cuvette standard」)には、cuvetteの吸光度測定値が表示されます(図 6)。Values'列には 280 nm での吸光度値が表示されます。
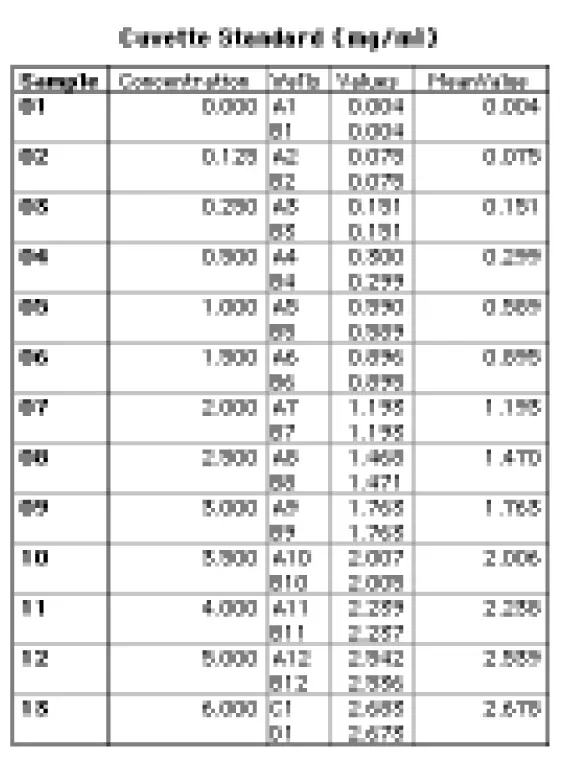
図 6:cuvetteセットのグループテーブル
次に、Plateとcuvetteからの標準曲線を 1 つのグラフにPlotしました。グラフセクションを作成し、Plotダイアログボックスを使用して、Groups ポップアップメニューから対応する Group table を選択し、各Plotの x 変数と y 変数を指定して 3 つのプロットを作成しました(図 7)。図に示す例では、ハイライトされたPlotは "Plate with PathCheck "です。データは "Plate Standard "グループに由来し、x 変数は濃度、y 変数は "MeanValue"(すなわち、パスレングス補正した吸光度の平均値)です。
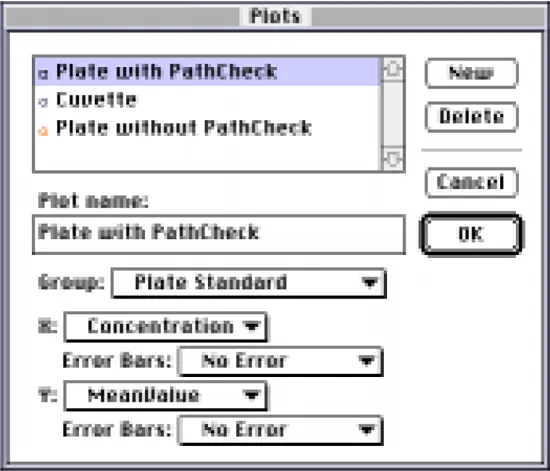
図7:グラフセクションに関連するPlotダイアログボックス
結果のグラフ(図 8)は、3 つのプロット(PathCheck の有無によるプレート値、cuvette値)を示しています。PathCheckなしの生のプレート吸光度のPlot(下線)は、ウェル内の容量(したがってウェル内の経路長)が均一でなかったため、不規則になっています。PathCheckを用いたプレート値のPlotは、最高濃度(6 mg/mL)まで直線的です。補正されたプレート値は、約3 mg/mlまではcuvette値と重ね合わせることができ、それ以上ではcuvetteのPlotが直線性から逸脱し始めます。補正されたプレートのプロットはcuvetteのPlotよりも上に広がっていますが、これは値が計算値であり、生の吸光度値(下のPlot)がまだ曲線の線形範囲内にあるためです。
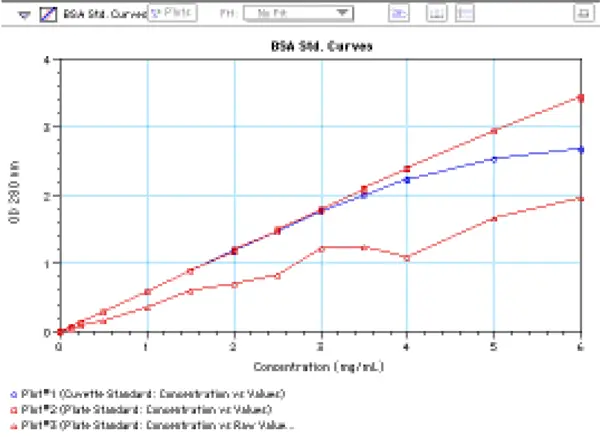
図 8:マイクロプレートとcuvetteで得られた BSA standard curve。標準物質は、1cm の光路長cuvette(❍)と、光路長補正あり(❒)と補正なし(❒)のマイクロプレートウェルで 280nmで読み取られました。
実施例2:ゲルろ過カラムでの抗体-酵素コンジュゲート精製時のカラム画分評価における PathCheck の使用
Pharmacia S-100 ゲルろ過カラムで抗体-酵素コンジュゲートを精製する際にフラクションを回収しました。ボイドボリュームのフラクションは廃棄し、フラクション21から61をそれぞれCostar UVプレートで280nmで読み取り、比較のために標準石英キュベットでも読み取りました。フラクション24から29は濃度が高すぎたため、吸光度が直線範囲になるように1対5に希釈しました。
グループテーブルの初期設定
SoftMax Proでは、希釈倍率はあるがstandard curveに対して定量する予定がないサンプル(カラム分画など)のグループテーブルを設定する方法が2つあります:
- グループに「Unknowns(希釈)」フォーマットを割り当てます。SoftMax Proは自動的に希釈倍率フィールドをアクティブにし、グループテーブルに希釈(倍率)とAdj.Resultの列を含めます。また、標準曲線が作成されることを想定し、今回の例(標準曲線に基づく濃度の計算と統計情報)には不要な5つの列が作成されます。これらの列は計算には必要ないので、グループ・テーブルから削除してもよいです。
- グループに'Basic'形式を割り当て、グループ設定ダイアログボックスまたは系列ダイアログボックスを使用して、希釈倍率情報を含むようにグループを修正します。1つは希釈倍率を含む列、もう1つは吸光度値に希釈倍率を乗じる列です。
この例では、2番目の方法でグループテーブルを作成し、ウェルに検体名を割り当てました。Plate Section Template Editorに "Plate "という新しいグループを作成し、Basic フォーマットを割り当てました。A~D行をハイライトし、SeriesボタンをクリックしてSeriesダイアログボックスにアクセスします(図9)
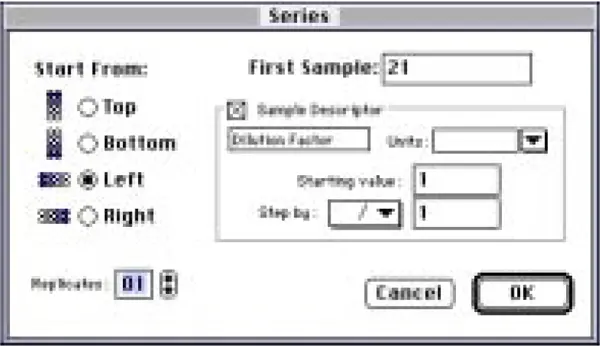
図9:カラム分画用に設定されたシリーズ設定ダイアログボックス
開始サンプルは21(分画番号に対応)に設定され、シリーズは左から右へ1レプリケートで進むように設定されました。Sample Descriptorボックスをチェックし、'dilution factor'を入力しました。単位フィールドはクリアして空白のままにしました。OKボタンをクリックしてシリーズを作成すると、ウェルに割り当てられたサンプル名(分画番号)がテンプレートに表示されました(図10)。希釈倍率が1以外のサンプルは、テンプレート内のウェルをクリックして選択し、希釈倍率フィールドに適切な倍率を入力して希釈倍率を追加しました。図では、ウェルA5(分画#25)がハイライトされ、そのサンプル名(25)と希釈倍率(5)がそれぞれのフィールドに表示されています。バッファーブランクはウェルD6とD7に割り当てられました。
注意:SoftMax Proのデフォルトでは行または列が完全に塗りつぶされるため、シリーズ機能は最初にD行すべてを塗りつぶしました。ウェルD6~D12は、系列がサンプル#61で停止するようにクリアされました。
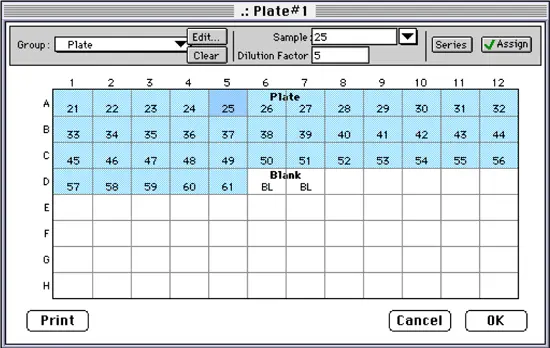
図10:プレート・セクションのテンプレート・エディター。
同様の方法で、同じサンプル名を CuvetteSet セクションに割り当てました。
実験の詳細とグループテーブルのカスタマイズ
装置の設定は、上記の実施例 1 で使用したものと同じです。プレートプリリード測定はCostar UVプレートで行いました。各カラム分画のアリコートとバッファーブランクを、使い捨てのトランスファーピペットを使用して指定のウェルに移し、SpectraMax PLUSで読み取りました。上記の例と同様に、PathCheck 機能の有用性を示すため、各ウェルの容量を意図的に変化させました。カラム分画も上記のように石英キュベットで読み取りました。
Plateセクションのグループ表(カスタマイズ後)を図11に示します。7つの列のうち、SoftMax Proが自動的に作成したのは3つの列(Sample、Wells、Correct.Values)のみです。(「Correct.Values」列はもともと「Values」というタイトルでしたが、この例では経路長補正値を生の吸光度値と区別するために名前を変更しました。他の4つのカラムにはカスタム式が与えられました。Fraction#"カラムは、サンプルに分画番号に対応する数値を与え、グラフ化できるように作成しました。(Sample列では、サンプル番号は名前として扱われるため、グラフ化することはできないです)。
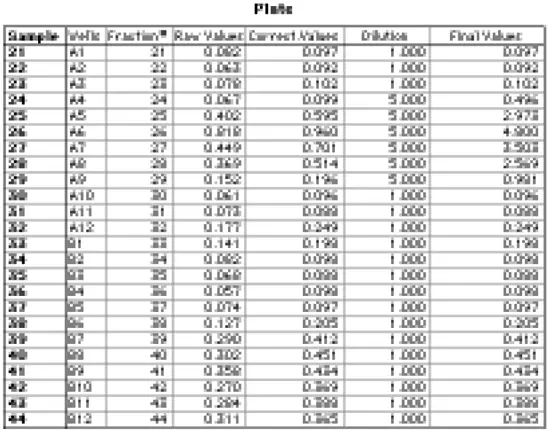
図11:マイクロプレートで読み取ったゲルろ過カラム分画のグループ表
分数#列のカスタム数式(図12)は、最初の項目が21となるように、"Index+20 "となっています。
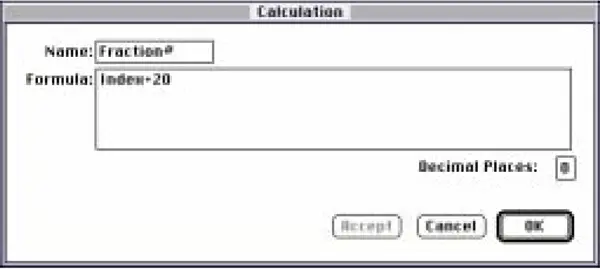
図12:「Fraction#」列の計算ダイアログボックス
formula: “Correct.Values*!WellPathLength” (Figure 13).
Raw Values "カラムには、経路長補正前の吸光度値が含まれ、カスタム式を用いて補正後の値から逆算して得られました: 「Correct.Values*!WellPathLength"(図13)
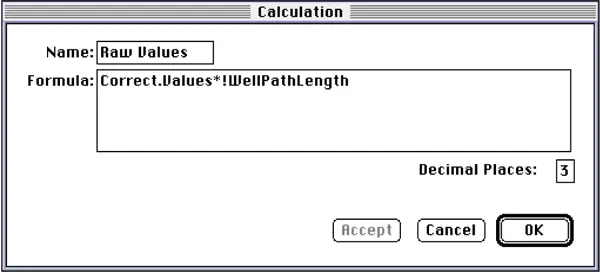
図13:「Raw Values」列の計算ダイアログボックス
「希釈」列のカスタム式を図14に示します。「因子」という用語は、テンプレートで定義されたサンプル記述子を指します。
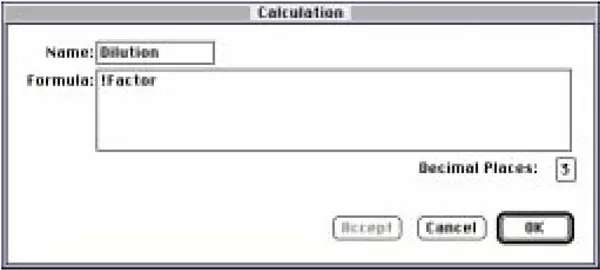
図14:「希釈」列の計算ダイアログボックス
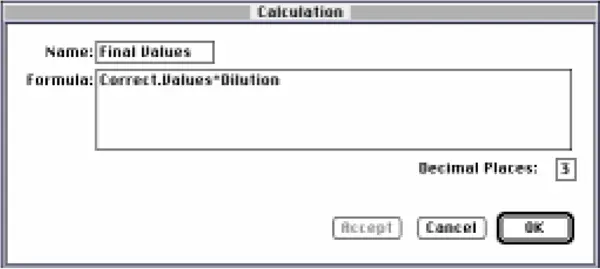
図15:「最終値#」列の計算ダイアログボックス
Final Values "列には、補正後の吸光度値に希釈倍率を乗じた値が表示されます。カスタム式は Correct.Values*Dilution です(図 15)。キュベットグループテーブルでは、吸光度値に希釈倍率を乗じるために同様のカラムを作成しました。
図 16 に、プレートとキュベットで得られたゲルカラムからの溶出プロファイルを示します。下側のプロット(プレートの値はパスレングス補正されていない)は、驚くことではないが、ウェル内の容量が均一でなかったため、上側のプロファイルを厳密に反映していません。しかし、プレートの値にPathCheckを適用すると、プレートのプロファイルは基本的にキュベットのプロファイルと重ね合わせることができました。
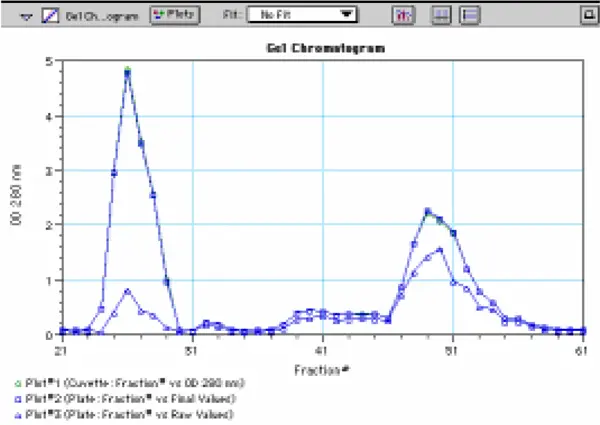
図16:抗体-酵素複合体の精製中に得られたカラム画分の溶出プロファイル。分画は、1 cm の光路長キュベット(❍)、および光路長補正を行った(❒)マイクロプレートウェル、および補正を行わなかった(∆)マイクロプレートウェルで 280 nm で読み取った。
上記の例は SpectraMax PLUS の PathCheck 機能の有用性を示しています。タンパク質溶液をマイクロプレートで読み取ることができ、吸光度値は標準的な1cmキュベットで同じ溶液を読み取った場合と同じです。正確なピペッティングは必要ありません。PathCheckのさらなる利点は、リニアレンジがキュベットのリニアレンジよりも拡大されることです(ウェルの生の吸光度値が装置のリニアレンジ内である限り)
マイクロプレートリーダーについて詳しくはこちら >>
PDF版(英語)