Application Note Quant-iT PicoGreen dsDNAアッセイキットを用いた
高感度なDNAの蛍光定量
- 50pg/mLまでのDNAを高感度に蛍光定量
- 4桁以上のリニアダイナミックレンジ
- SoftMax Proソフトウェアの設定済みプロトコールで結果を簡単に解析
PDF版(英語)
はじめに
Joyce Itatani | Application Scientist | モレキュラーデバイス
Cathy Olsen | Sr. Application Scientist | モレキュラーデバイス
二本鎖DNAは通常、吸光マイクロプレートリーダーでDNA溶液の吸光度を260 nmで測定することにより定量されます。しかし、この方法では、一般的な吸光マイクロプレートリーダーでは約250 ng/mLまでしか測定できません。次世代シーケンシングやDNA増幅産物の定量など、少量のサンプルを含む生物製剤のアプリケーションでは、より高感度な方法が必要とされます。サーモフィッシャーサイエンティフィック社のQuant-iT PicoGreen dsDNAアッセイキットはDNAに対する特異性が高く、従来の吸光度法の約1000倍の感度があります。製品マニュアルに記載されているように、マイクロプレートフォーマットでのこのアッセイのダイナミックレンジは、単一の色素濃度で250 pg/mLから1000 ng/mLです。ここでは、モレキュラーデバイス SpectraMax®マイクロプレートリーダーとQuant-iT PicoGreenアッセイを用いることで、50 pg/mLという低濃度の二本鎖DNAを確実に測定できることを実証します。
アッセイの感度を最大にするためには、最適なExcitation波長とEmission波長を使用する必要があります。フィルターベースのプレートリーダーとは異なり、SpectraMax iD5 マルチモードマイクロプレートリーダーやその他のSpectraMaxプレートリーダーのデュアルモノクロメーターでは、リーダーの指定範囲内で任意の波長を選択できます。フィルターベースプレートリー ダーやキットマニュアルで推奨されている波長とは多少異なる場合があるため、アッセイに最適な感度とダイナミックレンジを提供する励起および発光波長を決定することが重要です。
材料
- Quant-iT PicoGreen dsDNAアッセイキット(サーモフィッシャーサイエンティフィック)
- 黒色96ウェルマイクロプレート(Greiner Bio-One)
- ライトセーフブラックマイクロチューブ(Argos)
- マイクロプレートリーダー(注:ここに記載されていないSpectraMaxリーダーもPicoGreenアッセイに同様の性能があります。)
⚪︎SpectraMax Miniマルチモードマイクロプレートリーダー(モレキュラーデバイス P/N SMAX MINI AF)、蛍光フィルターキューブFL-535付き(モレキュラーデバイス P/N 5089097)
⚪︎SpectraMax® iD5マルチモードマイクロプレートリーダー(モレキュラーデバイス P/N #iD5)。
⚪︎SpectraMax® i3x マルチモードマイクロプレートリーダー(モレキュラーデバイス P/N #i3x)
⚪︎SpectraMax® M5マルチモードマイクロプレートリーダー(モレキュラーデバイス P/N #M5)
⚪︎SpectraMax® Gemini™ EMマイクロプレートリーダー(モレキュラーデバイス P/N #EM)
方法
装置とプロトコールのセットアップ
- マイクロプレートリーダーの電源を入れます。
- SoftMax® Proソフトウェアを起動し、Protocols dropdown menuからPicoGreen蛍光プロトコルを開きます。
使用するSpectraMaxリーダーによっては、アッセイに最適な設定を入力する必要があります(表1を参照)。 - [Settings(設定)]をクリックし、画面左側のオプションから[Wells to Read(読み取るウェル)]と[Assay Plate Type(アッセイプレートタイプ)]を選択します。
- Template(テンプレート)ボタンをクリックしてウィンドウを開き、マイクロプレートのウェル をあらかじめ設定されたtemplate groupに割り当てることができます。dropdown menuを使用して、適切なtemplate groupを選択します。PicoGreen 蛍光プロトコールには、Standards、Unknowns、Unknowns_NoDiln(希釈していないサンプルに使用)などのtemplate groupがあらかじめ設定されています。あらかじめ設定されたtemplate groupにウェルを割り当てることで、プロトコルのgroup tablesにマイクロプレート読み取り時に取得される対応するデータが入力されます。
アッセイの準備
このアッセイ方法は、QuantiT PicoGreen dsDNA 試薬とキットの製品情報シートに記載されている手順に従いますが、96ウェルマイクロプレートフォーマットに適合するようにアッセイ容量を2.0mLから200μLに減らします。
- TEバッファー中の濃縮DMSO溶液を200倍に希釈して、Quant-iT PicoGreen試薬の水性作業溶液を調製します(上記で調製)。
- 試薬がガラス表面に吸着する可能性があるため、ガラスではなくプラスチック容器での調製を推奨します。茶色または黒色のチューブを使用するか、ホイルで覆うなどして、溶液を光から保護します。この溶液は調製後数時間以内に使用します。
- DNA 標準曲線: dsDNAの2μg/mLストック溶液をTEで調製します。キットに付属のλDNAスタンダードをTEで50倍に希釈して2μg/mL溶液にします。
注:場合によっては、アッセイするDNAの種類に類似したDNAを用いてstandard curveを作成することが望ましい場合があります。
| Parameter | SpectraMax iD5/iD3 | SpectraMax i3x | |||
|---|---|---|---|---|---|
| Read mode | Fluorescence (FL) | Fluorescence (FL) | Fluorescence (FL) | Fluorescence (FL) | Fluorescence (FL) |
| Read type | Endpoint | Endpoint | Endpoint | Endpoint | Endpoint |
| Wavelengths | Excitation: 485 nm Emission: 535 nm |
Excitation: 490 nm bandwidth 9 nm Emission: 525 nm bandwidth 15 nm |
Excitation: 490 nm Emission: 525 nm Emission cutoff: 515 nm |
Excitation: 490 nm Emission: 525 nm Emission cutoff: 515 nm |
Fluorescence filter cube: FL-535 (Excitation 485 nm, Emission 535 nm, Dichroic 508 nm) |
| Plate type and Read area | Select based on microplate and wells used | Select based on microplate and wells used | Select based on microplate and wells used | Select based on microplate and wells used | Select based on microplate and wells used |
| PMT and Optics | PMT gain: Automatic Integration time: 200 ms Read height: Optimize for microplate used |
PMT gain: N/A Flashes per read: 10 Read height: Optimize for microplate used |
PMT gain: Automatic Flashes per read: 10 |
PMT gain: Automatic Flashes per read: 10 |
PMT gain: Automatic Integration time: 400 ms Read height: Optimize for microplate used |
表1. SpectraMaxプレートリーダーの装置設定。SpectraMax iD5、i3x、Miniプレートリーダーの場合、read height設定は使用するマイクロプレートに合わせて最適化する必要があります。注:同様の性能を持つ他のプレートリーダーもリストアップしています。
- 必要であれば、1ng/mLから1μg/mLまでのハイレンジ標準曲線を作成しても、25pg/mLから25ng/mLまでのローレンジ標準曲線を作成しても良いです。ハイレンジ曲線またはローレンジ曲線では、1:10希釈液を使用することができます。低レンジ曲線では、2 μg/mL の溶液を 40 倍に希釈し、50 ng/mL の出発溶液を調製します。
- このアプリケーションノートでは、50 pg/mL から 1 μg/mL までの一連の標準液を 1:3 希釈系列として設定しました。
標準液を黒色96ウェルマイクロプレートに1ウェル当たり100μLずつ、できればトリプリケートでピペッティングします。TEのみを封じ込めた(DNAを封じ込めなかった)バッファーブランクウエルも必ず含めます。 - 各ウェルにQuantiT PicoGreen試薬の水溶液を100μLずつ加えます(この結果、ウェル内の標準液を1:2に希釈することになります)。トリチュレーションまたはプレートシェーカーでよく混ぜ、遮光して室温で 2~5 分間インキュベートします。
マイクロプレートの読み取り
- SpectraMax Mシリーズプレートリーダーを使用する場合は、紫色のプレートアダプターが マイクロプレートリーダーの引き出しにあることを確認します。マイクロプレートをドロワーにセットします。
- SoftMax® ProソフトウェアのReadボタンをクリックします。
データの分析
- マイクロプレートの読み取りが完了すると、相対蛍光単位(RFU)がPlateセクションに表示されます。データは、テンプレートのセットアップ時に作成されたGroup Tablesで分析されます。Group Tableの代表的なデータの例については、表2を参照してください。
- Templateで割り当てられた(したがってStandards group tableに表示された)標準物質は、プロトコルのStandard Curveセクションに自動的にplotされます。デフォルトではlinear curve fitが適用されますが、wide dynamic rangeのstandard curveをplotする場合はlog-log fitを使用することもできます。Curve fitは、graph sectionのdropdown Fit menuからセレクトします。
| Sample | DNA conc (ng/mL) | Average RFU | StdDev | %CV |
|---|---|---|---|---|
| 1 | 1000.00 | 54893498 | 611149.3 | 1.1 |
| 2 | 333.333 | 17114392 | 685401.0 | 4.0 |
| 3 | 111.111 | 5884808 | 249140.5 | 4.2 |
| 4 | 37.037 | 1938706 | 21246.0 | 1.1 |
| 5 | 12.346 | 667849 | 10336.2 | 1.5 |
| 6 | 4.115 | 231812 | 9389.3 | 4.1 |
| 7 | 1.372 | 74577 | 4324.4 | 5.8 |
| 8 | 0.457 | 25963 | 2403.5 | 9.3 |
| 9 | 0.152 | 8981 | 1422.3 | 15.8 |
| 10 | 0.051 | 2441 | 1205.6 | 49.4 |
表2. DNAスタンダード。
結果
Quant-iT PicoGreen dsDNAアッセイキットとSpectraMaxプレートリーダーを用いて、50 pg/mL~1 μg/mLのDNAスタンダードを検出しました(SpectraMax iD5プレートリーダーのデータを示すが、他のSpectraMaxプレートリーダーでも同様の結果が得られました)。SoftMax® Proソフトウェアは、標準反復の各セットの平均RFU、標準偏差、%CVを自動的に算出しました。SoftMax Proソフトウェアのlog-log curve fitを用いてstandard curveをplotした(図1)。96ウェルマイクロプレートフォーマットで50 pg/mLまでの感度が観察され、標準検出限界はブランクの標準偏差の3倍であった。これはQuant-iT PicoGreenアッセイの製品添付文書に記載されている検出下限250pg/mLを大きく下回っています。図2はhigh-range(A)とlow-range(B)のstandard curveを示しています。直線性は、標準物質の範囲全体にわたって優れていました(各曲線については \( r^2 \geq 0.99 \) )。
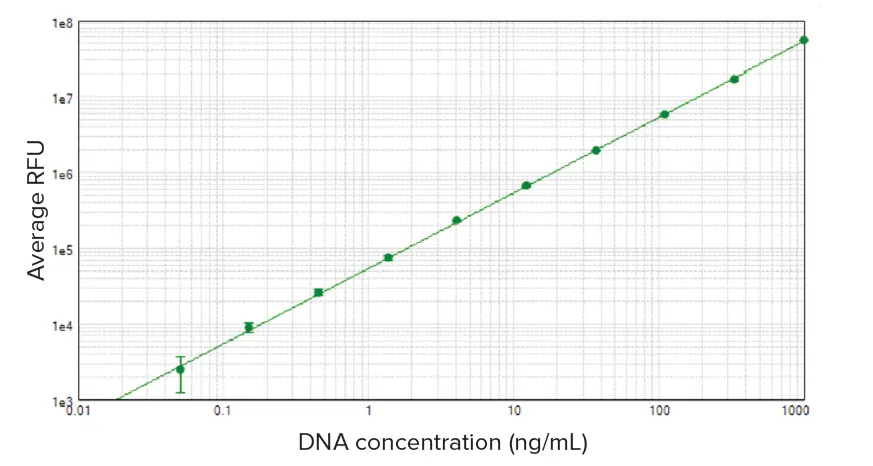
図1. DNA standard curve。50pg/mLから1000ng/mLまでのDNA standardをQuant-iT PicoGreen dsDNAアッセイキットを用いてSpectraMax iD5プレートリーダーでアッセイしました。 standard curveはSoftMax Proソフトウェアのlog-log curve fitを用いてplotした( \( r^2 \geq 1.00 \) )。
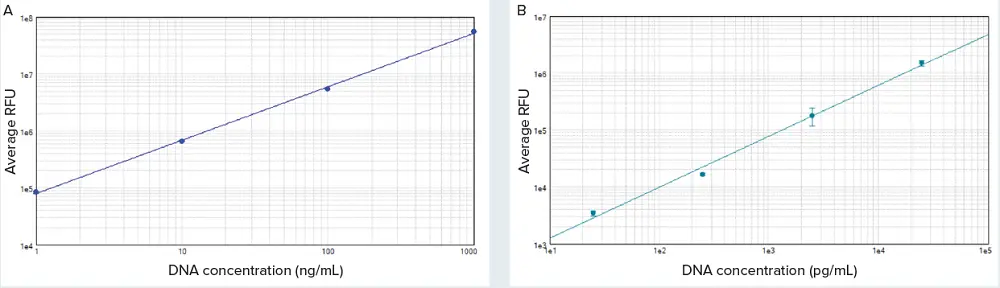
図2. High-range(A)とlow-range(B)のstandard curve。curveはSoftMax® Proソフトウェアのlog-log curve fitを用いてplotした(両曲線は、 \( r^2 \geq 0.99 \) )。
結論
Quant-iT PicoGreen dsDNAアッセイキットをSpectraMaxマイクロプレートリーダーとSoftMax Proソフトウェアで実行すると、二本鎖DNAを迅速かつ高感度に検出できます。ソフトウェアの分析機能により、読みやすく、ユーザーがカスタマイズ可能なレポート形式で定量を行うことができます。ソフトウェアには設定済みのプロトコールが用意されており、迅速なアッセイセットアップが可能です。
PDF版(英語)