Application Note Quant-iT RiboGreenでRNAを高感度蛍光定量
- RNAを100 pg/mLまで高感度に蛍光定量します
- 4桁の線形ダイナミックレンジをカバーします
- SoftMax Proソフトウェアの事前設定済みプロトコールで、データ取得と解析を簡単に行えます
PDF版(英語)
Beverly Pappas, PhD | BioResearch Field Applications Scientist|モレキュラーデバイス
はじめに
核酸濃度の正確な定量は、トランスフェクション、クローニング、PCR、次世代シーケンシング(NGS)などの後続アプリケーションにとって重要です。これらのアプリケーションでは、最適な性能を発揮するために特定の核酸濃度が必要になることがあります。不正確な定量は、後続アッセイのばらつきを増加させ、結果の品質に影響を与える可能性があります *1。
RNAは通常、マイクロプレートリーダーで260 nmの吸光度を測定することで定量されます。しかし、吸光度ベースの方法には、コンタミネーションの影響、非特異的タンパク質や遊離ヌクレオチドからのシグナル寄与、感度の限界などの欠点があります *2。Thermo Fisher Scientific社のQuant-iT™ RiboGreen RNAアッセイキットは、RNA定量により特異的です。製品マニュアルによると、このアッセイのマイクロプレートフォーマットでのダイナミックレンジは、2種類の染料濃度を使用して1 ng/mLから1 μg/mLであり、吸光度法より約1000倍高感度です *3。本アプリケーションノートでは、SpectraMax® iD5 マルチモードマイクロプレートリーダーを使用し、Quant-iT RiboGreenアッセイでRNA濃度を100 pg/mLまで確実に測定できることを示します。他のSpectraMaxプレートリーダーでも同様の結果が得られました。
アッセイの感度を最大化するため、SpectraMax iD5およびSpectraMax® i3x マルチモードマイクロプレートリーダーで取得したリードにはSpectral Optimization Wizardを使用し、最適な励起波長と蛍光波長を決定しました。このウィザードを使用することで、SpectraMax iD5およびi3xプレートリーダーのデュアルモノクロメーターは最適な波長ペアに調整され、アッセイに対して最高の感度とダイナミックレンジを提供します。
材料
・Quant-iT RiboGreen RNA アッセイキット(Invitrogen)
・96ウェル黒色マイクロプレート(Greiner Bio-One)
・ライトセーフブラックマイクロチューブ (Argos)
・マイクロプレートリーダー
SpectraMax iD5 マルチモードマイクロプレートリーダー(モレキュラーデバイス P/N iD5)
SpectraMax i3x マルチモードマイクロプレートリーダー(モレキュラーデバイス P/N i3x)
SpectraMax® M5e マルチモードマイクロプレートリーダー(モレキュラーデバイス P/N M5e)
蛍光フィルターキューブFL-535付きSpectraMax® Miniマルチモードマイクロプレートリーダー(モレキュラーデバイス P/N SMAX MINI AF)(モレキュラーデバイス P/N 5089097)
方法
装置とプロトコールの設定
- マイクロプレートリーダーの電源を入れます。
- SoftMax® Pro ソフトウェアを起動し、Protocol ManagerのProtocol LibraryドロップダウンメニューからNucleic Acidsフォルダー内のRiboGreen Fluorescenceプロトコールを開きます。使用するSpectraMaxプレートリーダーに応じて、アッセイに最適化された設定を選択します(表1参照)。
- 「Acquisition Settings」をクリックし、画面左側のオプションから読み取るウェルとアッセイプレートタイプを選択します。
- 「Template Editor」ボタンをクリックし、マイクロプレートのウェルをプリセットテンプレートグループに割り当てます。RiboGreen Fluorescenceプロトコールには、Standards、Unknowns、Unknowns_Diln(希釈サンプル用)などの事前設定済みテンプレートグループがあります。Addを選択して、High-Range StandardsやLow-Range Standardsのユニークなグループを追加できます。
アッセイの準備
以下の方法は、Quant-iT RiboGreen RNA試薬およびキットの製品情報シートに記載された手順に従いますが、アッセイ容量は96ウェルマイクロプレートフォーマットに合わせて2.0 mLから200 μLに比例縮小しています。
- 1X TEバッファー(10 mM Tris-HCl、1 mM EDTA、pH 7.5)を、キットの濃縮バッファーを蒸留DEPC処理水で20倍に希釈して調製します。
- Quant-iT RiboGreen試薬の水性ワーキング溶液を、濃縮DMSO溶液を1X TEバッファーで200倍希釈(高レンジアッセイ用:20 ng/mL~1 μg/mL RNA)および2000倍希釈(低レンジアッセイ用:1 ng/mL~50 ng/mL RNA)して調製します。ガラス表面への吸着を避けるため、15 mLコニカルチューブで調製します。溶液はアルミホイルで覆い、光を避けて保護します。
- 高レンジRNA標準曲線:TE中で2 μg/mLのRNAストック溶液を調製します。キット付属のラムダRNA標準をTEで50倍希釈して2 μg/mL溶液を作成します。本アプリケーションノートでは、20 ng/mL~1 μg/mLの標準系列をLight-Safe黒色マイクロチューブで調製しました(表2参照)。
- 低レンジRNA標準曲線:TE中で100 ng/mLのRNAストック溶液を調製します。2 μg/mL RNAストック溶液をTEで20倍希釈して100 ng/mL溶液を作成します。本アプリケーションノートでは、0.0008 ng/mL~25 ng/mLの標準系列を表3aおよび3bに従って調製しました。表3aはRNAseフリーのマイクロチューブで低レンジ希釈を得るための段階希釈を示し、表3bはLight-Safe黒色マイクロチューブで低レンジ希釈に2000倍希釈したQuant-iT RiboGreen試薬500 μLを添加した後の最終RNA濃度を示します。この段階的アプローチは、SpectraMaxプレートリーダーでの検出下限を、キットの感度1 ng/mLと比較するために実施しました。
- ワーキング溶液中の標準液を、固体黒色96ウェルマイクロプレートに各ウェル200 μLずつ、3連で分注します。TEのみ(RNAなし)のブランクウェルも3連で含めます。
- 室温で2分間、光を避けてインキュベートします。
マイクロプレートの読み取り
- マイクロプレートをドロワーにセットします。SpectraMax Mシリーズプレートリーダーの場合、プレートキャリッジの紫色プレートアダプターの上にマイクロプレートを置きます。
- SoftMax ProソフトウェアのでReadボタンをクリックします。
データ解析
- マイクロプレートの読み取り後、ブランクグループは自動的にデータから差し引かれ、補正済みの相対蛍光単位(RFU)がPlateセクションに表示されます。データはテンプレート設定時に作成されたGroup Tablesで解析されます。
- 各プレートリーダーで読み取った各標準の補正RFUは、最高標準(1 μg/mL)の値に正規化され、High-Range Standard CurveおよびLow-Range Standard Curveセクションにプロットされます。グラフセクションのドロップダウンメニューから線形カーブフィットが選択されます。
| Parameter | SpectraMax iD5/iD3 | SpectraMax i3x | SpectraMax Mini | |
|---|---|---|---|---|
| Read mode | Fluorescence (FL) | Fluorescence (FL) | Fluorescence (FL) | Fluorescence (FL) |
| Read type | Endpoint | Endpoint | Endpoint | Endpoint |
| Wavelengths |
Excitation: 480 Emission: 520 |
Excitation: 480 Emission: 520 |
Excitation: 480 Emission: 525 Emission Cutoff: 515 |
FL-535 filter cube Excitation: 485/20 Emission: 535/25 |
| Plate type and read area | Select based on microplate and wells used | Select based on microplate and wells used | Select based on microplate and wells used | Select based on microplate and wells used |
| PMT and optics | PMT gain: Automatic Integration time: 200 ms Read height: optimize for microplate used | PMT gain: Automatic Integration time: 200 ms Read height: optimize for microplate used | PMT gain: Automatic Flashes per read: 6 | PMT gain: Automatic Integration time: 200 ms Read height: optimize for microplate used |
表1. SpectraMaxプレートリーダーの装置設定 SpectraMax iD5、i3x、およびMiniプレートリーダーでは、使用するマイクロプレートに合わせてリード高さ設定を最適化する必要があります。注記:同等の性能を持つ追加のプレートリーダーも記載されています。
| Standard | Volume (μL) of 1X TE | Volume (μL) of \( \frac{2\ \text{μg}}{\text{mL}} \) RNA Stock | Volume (μL) of 200-fold diluted Quant-iT™ RiboGreen Reagen | Final RNA concentration in Quant-iT™ RiboGreen Assay |
|---|---|---|---|---|
| High-Range Standard 1 | 0 | 500 | 500 | \( \frac{1\ \text{μg}}{\text{mL}} \) |
| High-Range Standard 2 | 250 | 250 | 500 | \( \frac{500\ \text{ng}}{\text{mL}} \) |
| High-Range Standard 3 | 450 | 50 | 500 | \( \frac{100\ \text{ng}}{\text{mL}} \) |
| High-Range Standard 4 | 490 | 10 | 500 | \( \frac{20\ \text{ng}}{\text{mL}} \) |
| High-Range Blank | 500 | 0 | 500 | 0 |
表2. 高レンジ標準曲線の調製プロトコール
| Volume (μL) of 1X TE | Volume (μL) RNA | Working RNA Concentration (ng/mL) | |
|---|---|---|---|
| Dilution 1 | 0 | 1000 μL of 100 ng/mL RNA | 50 |
| Dilution 2 | 500 | 500 μL of Dilution 1 | 25 |
| Dilution 3 | 800 | 200 μL of Dilution 2 | 5 |
| Dilution 4 | 800 | 200 μL of Dilution 3 | 1 |
| Dilution 5 | 800 | 200 μL of Dilution 4 | 0.2 |
| Dilution 6 | 800 | 200 μL of Dilution 5 | 0.04 |
| Dilution 7 | 800 | 200 μL of Dilution 6 | 0.008 |
| Dilution 8 | 800 | 200 μL of Dilution 7 | 0.0016 |
| Dilution 9 | 600 | 0 | 0 |
表3a. 低レンジ希釈の調製プロトコール。1X TEと100 ng/mL RNAストック溶液を使用して段階的にRNA希釈を行います。
| Low-Range Dilution | Volume (μL) of 2000-fold Diluted Quant-iT™ RiboGreen® Reagent | Final RNA Concentration in Quant-iT™ RiboGreen® Assay (ng/mL) | |
|---|---|---|---|
| Low-Range Standard 1 | 500 μL of Dilution 1 | 500 | 25 |
| Low-Range Standard 2 | 500 μL of Dilution 2 | 500 | 12.5 |
| Low-Range Standard 3 | 500 μL of Dilution 3 | 500 | 2.5 |
| Low-Range Standard 4 | 500 μL of Dilution 4 | 500 | 0.5 |
| Low-Range Standard 5 | 500 μL of Dilution 5 | 500 | 0.1 |
| Low-Range Standard 6 | 500 μL of Dilution 6 | 500 | 0.020 |
| Low-Range Standard 7 | 500 μL of Dilution 7 | 500 | 0.004 |
| Low-Range Standard 8 | 500 μL of Dilution 8 | 500 | 0.0008 |
| Low-Range Blank | 500 μL of Dilution 9 | 500 | 0 |
表3b. 低レンジ標準曲線の調製プロトコール。表3aで得られた低レンジ希釈に、2000倍に希釈したQuant-iT RiboGreen試薬を添加し、最終RNA濃度を得ます。
結果
Quant-iT RiboGreenアッセイキットとSpectraMaxプレートリーダーを使用して、100 pg/mLから1 μg/mLまでのRNA標準を検出しました。RiboGreen蛍光プロトコールでは、SoftMax Proソフトウェアが各標準の平均RFU、標準偏差、および%CVを自動的に計算しました。標準曲線は、SpectraMax iD5、i3x、M5e、およびMiniプレートリーダーで実行された高レンジおよび低レンジ標準に対して、SoftMax Proソフトウェアの線形カーブフィットを使用してプロットされました(図1Aおよび1B)。各曲線の線形性は非常に良好でした(r² ≥ 0.990)。
96ウェルマイクロプレートフォーマットを使用し、低レンジブランクの標準偏差の3倍を検出限界とする標準計算により、100 pg/mLまでの感度が確認されました。これは、Quant-iT RiboGreenアッセイ製品インサートに記載されている1 ng/mLより10倍低く、感度が成功の鍵となるアプリケーションにおけるアッセイの有用性を高めることを示しています。
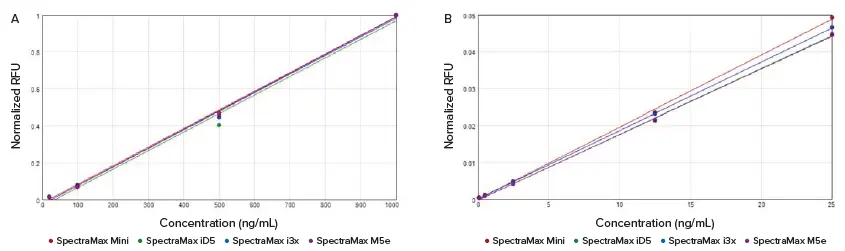
図1. 4種類のSpectraMaxプレートリーダーで実行された高レンジ(A)および低レンジ(B)の正規化標準曲線。 各標準の補正平均RFUは、テストしたプレートリーダーで最高標準RFU(1 μg/mL)に正規化されました。曲線はSoftMax Proソフトウェアの線形カーブフィットを使用してプロットされました(高レンジ曲線 r² ≥ 0.990、低レンジ曲線 r² ≥ 0.999)。
結論
Quant-iT RiboGreen RNAアッセイキットは、蛍光検出モード付きSpectraMaxマイクロプレートリーダーとSoftMax Proソフトウェアで実行すると、RNAの迅速で高感度な検出法となります。ソフトウエアの分析機能により、読みやすく、ユーザーがカスタマイズできるレポート形式で自動定量が可能です。
参考文献
- https://www.sigmaaldrich.com/US/en/technical-documents/technical-article/genomics/dna-and-rna-purification/dna-rna-quantification
- https://www.promega.com/resources/pubhub/choosing-the-right-method-for-nucleic-acid-quantitation/#references-c9a7b496-4311-4517-8968- f79b4eb55b34
- https://www.eppendorf.com/product-media/doc/en/59777/Eppendorf_Detection_Application-Note_271_Eppendorf-BioSpectrometer-fluorescence_ Determination-nucleic-acid-concentrations-fluorescent-dyes-Eppendorf-BioSpectrometer-fluorescence.pdf
PDF版(英語)